6 Visualización de datos usando ggplot2
Para comenzar, primero debemos importar el o los paquetes requeridos. ggplot2 está incluido en el paquete tidyverse.
library(tidyverse)En caso de que la tabla de datos de estudio no esté en la memoria de R, puedes cargarla nuevamente.
data <- read_csv("data/IR_table1.csv")ggplot2 es un paquete para graficar, que facilita crear gráficas complejas a partir de datos en un data frame. Incluye varias funciones para especificar que variables graficar, como éstas son expuestas, y varias otras características visuales. ggplot2 funciona mejor con datos extensos, i.e., una columna por cada dimensión, y una fila por cada observación. Tener datos bien estructurados te ayudará a crear figuras más eficientemente.
6.1 Graficar con ggplot2
Las gráficas de ggplot son construidas paso a paso, agregando nuevos elementos cada vez. Para graficar con ggplot, vamos a usar una línea de comando templado que es útil para diferentes tipos de gráficos:
ggplot(data = , mapping = aes()) + ()Usamos la función
ggplot()y el argumentodatapara indicar a partir de qué datos se debe crear la gráfica. Luego, la funciónaes()(aesthetic) para seleccionar las variables a graficar y como presentarlas, e.g. ejes x e y o características como tamaño, forma, color, etc.ggplot2ofrece “geoms” para indicar la representación gráfica de los datos (puntos, líneas, barras), algunos de ellos son:geom_point()para gráficos de dispersión.geom_boxplot()para gráficos de caja (boxplots).geom_line()para líneas de tendencia, series de tiempo, etc.
Usa + para agregar un geom a línea de comando de ggplot.

Notas
- Toda la información o inputs que agregues en la función
ggplot()son considerados por cualquiergeom(i.e., éstas son configuraciones universales), lo que incluye las variables para el eje x e y indicados usandoaes(). - También puedes especificar inputs para un
geomdado, independientemente de los definidos en la funciónggplot(). - El signo
+se usa para añadir nuevas funciones y se usa al final de cada línea que contiene la función anterior.
6.2 Construyendo gráficos iterativamente
Construir gráficos con ggplot2 es comúnmente un proceso iterativo. Primero definimos un set de datos a utilizar, luego indicamos las variables a graficar (ejes x e y), y escogemos un geom.

Seguidamente, empezamos a modificar el gráfico para extraer más información de él y hacerlo más explíito o auto-explicativo. Por ejemplo, podemos agregar transparencia (alpha) para evidenciar la sobre-posición de los puntos (overplotting). También podemos colorear los puntos (color).

Además, es posible “anotar” tus datos de acuerdo a cierta(s) variable(s) categórica(s), es decir, colorearlos o asignarles formas según ciertas características que tu consideres influyentes en tus datos. Por ejemplo, vamos a anotar nuestra gráfica por especie (“species”) y por locación geográfica (“geo_loc_name”).
ggplot(data = data, aes(x = observed, y = shannon)) + geom_point(alpha = 0.7, aes(color = geo_loc_name, shape = species))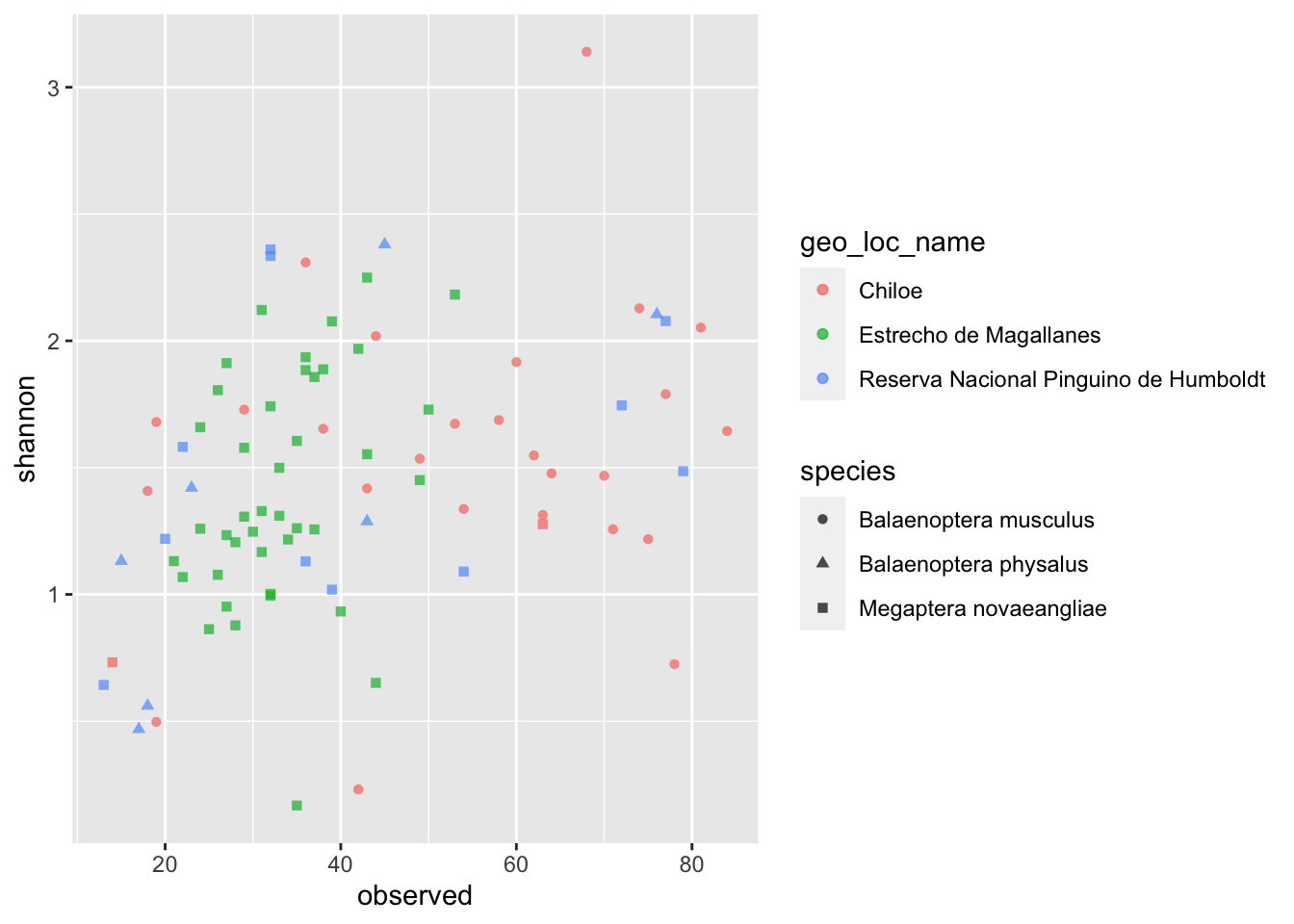
6.3 Boxplot
Boxplots o gráficos de caja, son útiles para visualizar la distribución de los datos de acuerdo a una variable o condición de interés.
- Por ejemplo, para visualizar la distribución de número de taxas observadas en el microbioma de la piel de las ballenas muestreadas en Chiloé, Estrecho Magallanes y Parque Nacional Pingüino de Humboldt:

- Sin embargo, ¿podría éste resultado estar sesgado por el tamaño muestreal de cada locación geográfica?. Veamos cuántas muestras tenemos por locación geográfica:
- Agregar puntos al boxplot nos da una mejor idea del número de muestras y su distribución:
ggplot(data = data, aes(x = geo_loc_name, y = observed)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5, color = "tomato")
¿Notas como las cajas (boxplot layer) están detrás de los puntos (jitter layer)? ¿Qué necesitas cambiar en el código de ggplot para que las cajas aparezcan en frente de los puntos? (Pista: el orden sí importa)
6.4 Graficar datos con series de tiempo
Cuando tienes datos tomados en una serie de tiempo, una buena manera de visualizarlos es a través de un gráfico de líneas. En el siguiente ejemplo vamos a utilizar una tabla de datos correspondiente a un sub-muestreo de un experimento RNAseq, que contiene las read counts de 8 transcritos después de 0, 3, 6, 12 y 24 horas de exposición a un estímulo X.
- Primero vamos a cargar dicha tabla a la memoria de R:
## Parsed with column specification:
## cols(
## transcripts = col_character(),
## `0hrs` = col_double(),
## `3hrs` = col_double(),
## `6hrs` = col_double(),
## `12hrs` = col_double(),
## `24hrs` = col_double()
## )# Usamos la función read_table2() para leer el archivo `data/VD_table2.tbl` y asignarlo al nuevo objeto `tr_counts`.
# El argumento col_names = TRUE es para indicar la presencia de títulos o nombres para cada columna.
tr_counts # dale un vistazo a la nueva tabla de datos## # A tibble: 8 x 6
## transcripts `0hrs` `3hrs` `6hrs` `12hrs` `24hrs`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Transcript_1 2.67 24.5 59.2 9.00 14.2
## 2 Transcript_2 0 87.0 5.71 7.61 0
## 3 Transcript_3 1.84 54.9 131. 63.5 15.6
## 4 Transcript_4 0.256 16.6 27.1 1.33 18.5
## 5 Transcript_5 0 0 0 0 0
## 6 Transcript_6 0 6.31 0 0 0
## 7 Transcript_7 0 6.64 0 0 0
## 8 Transcript_8 0 767. 92.7 141. 0Generalmente, a través de la metodología de RNAseq, podemos obtener una tabla como tr_counts, que informa de las read counts de cada transcrito por tiempo muestral. Sin embargo, para crear un gráfico lineal, usando ggplot() + geom_line(), en el que el eje X represente una serie de tiempo, es necesario que los tiempos muestreales sean una variable (no nombres de columnas, como en tr_counts).
- Entonces, recordamos lo aprendido anteriormente y utilizamos la función
gather()para remodelartr_counts, reuniendo los nombres de las columnas (tiempos) y transformándolos en un set de variables:
- Ahora podemos graficar las read counts de cada transcrito a través de los 5 puntos muestreales, usando
ggplot() + geom_line():
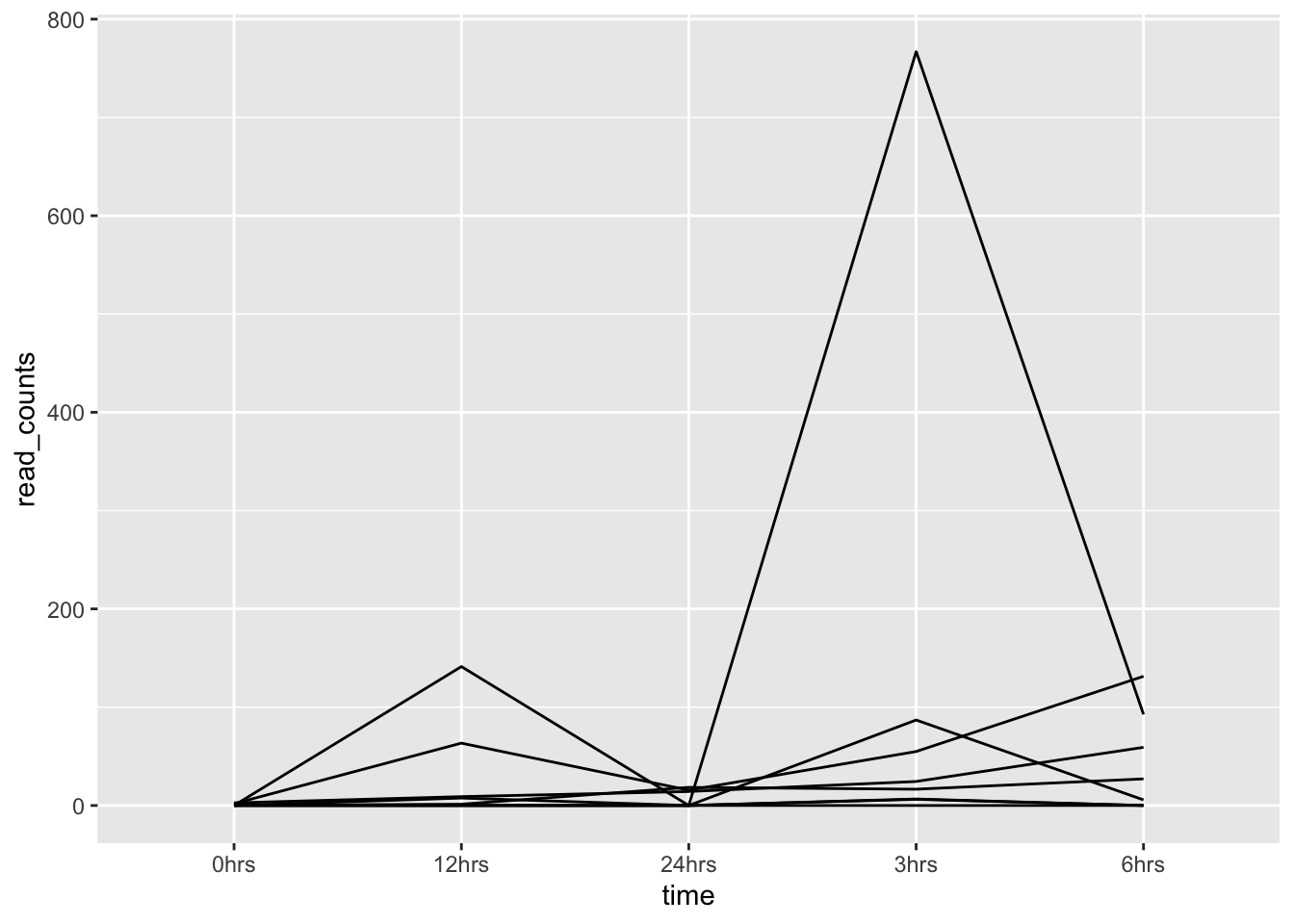
- ¿Aún no? Podemos anotar las líneas para identificar cada transcrito:
ggplot(data = tr_plot, aes(x = time, y = read_counts, group = transcripts)) +
geom_line(aes(color = transcripts))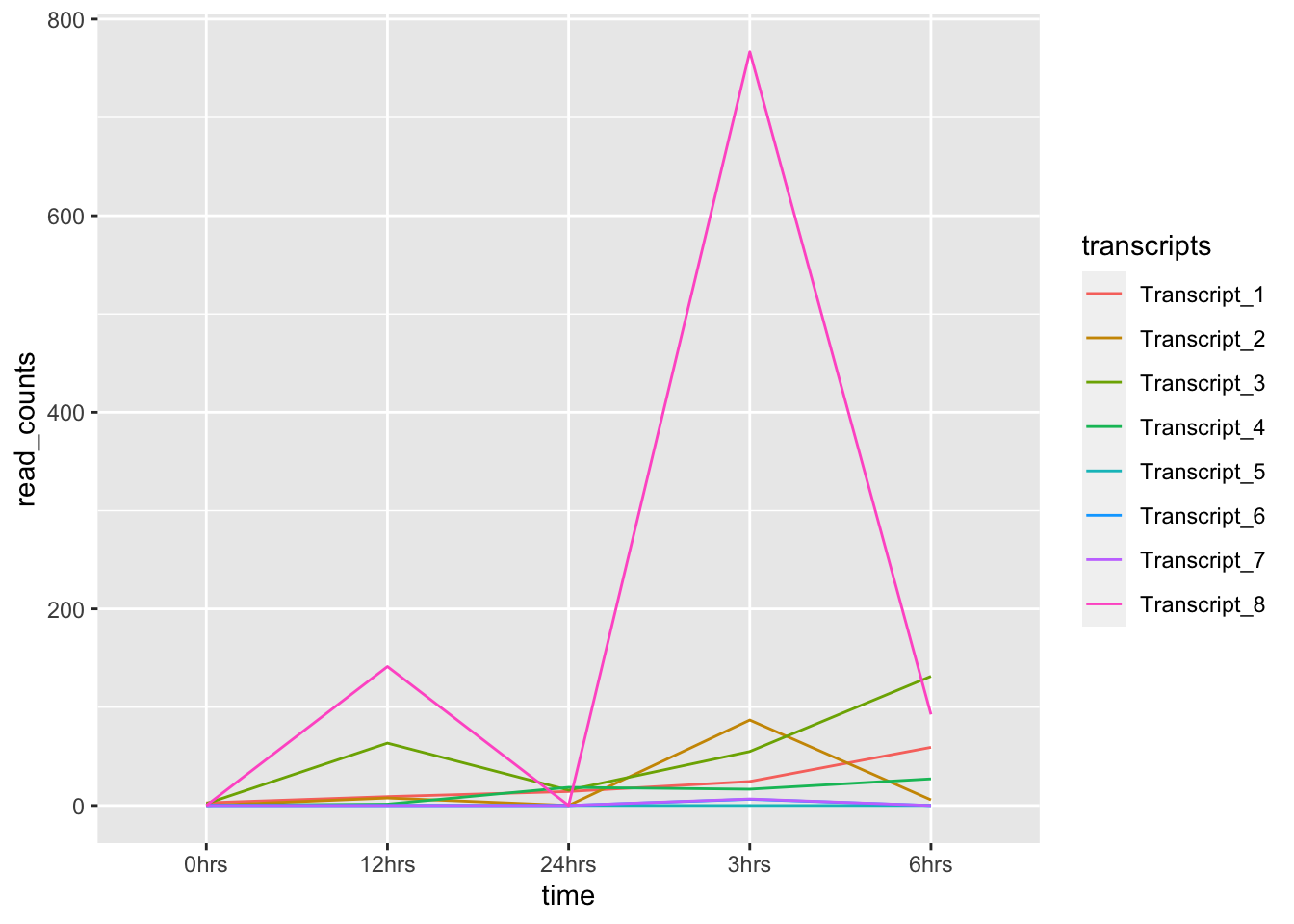
Como ya habrás notado, tanto en la gráfica como en la tabla, el rango de los valores de read counts es bastante amplio…
## [1] 0## [1] 766.8096Al graficar, en muchos cachos es conveniente considerar la escala de las observaciones, con el fin de obtener una mejor visualización de la distribución de los datos.
- Vamos a convertir los valores de read counts al logarítmo base 10 de las read counts, de modo de reducir el rango de distribución de los valores. Para ello, modificamos la escala del eje Y usando
scale_y_log10():
ggplot(data = tr_plot, aes(x = time, y = read_counts, group = transcripts)) +
geom_line(aes(color = transcripts)) +
scale_y_log10()## Warning: Transformation introduced infinite values in continuous y-axis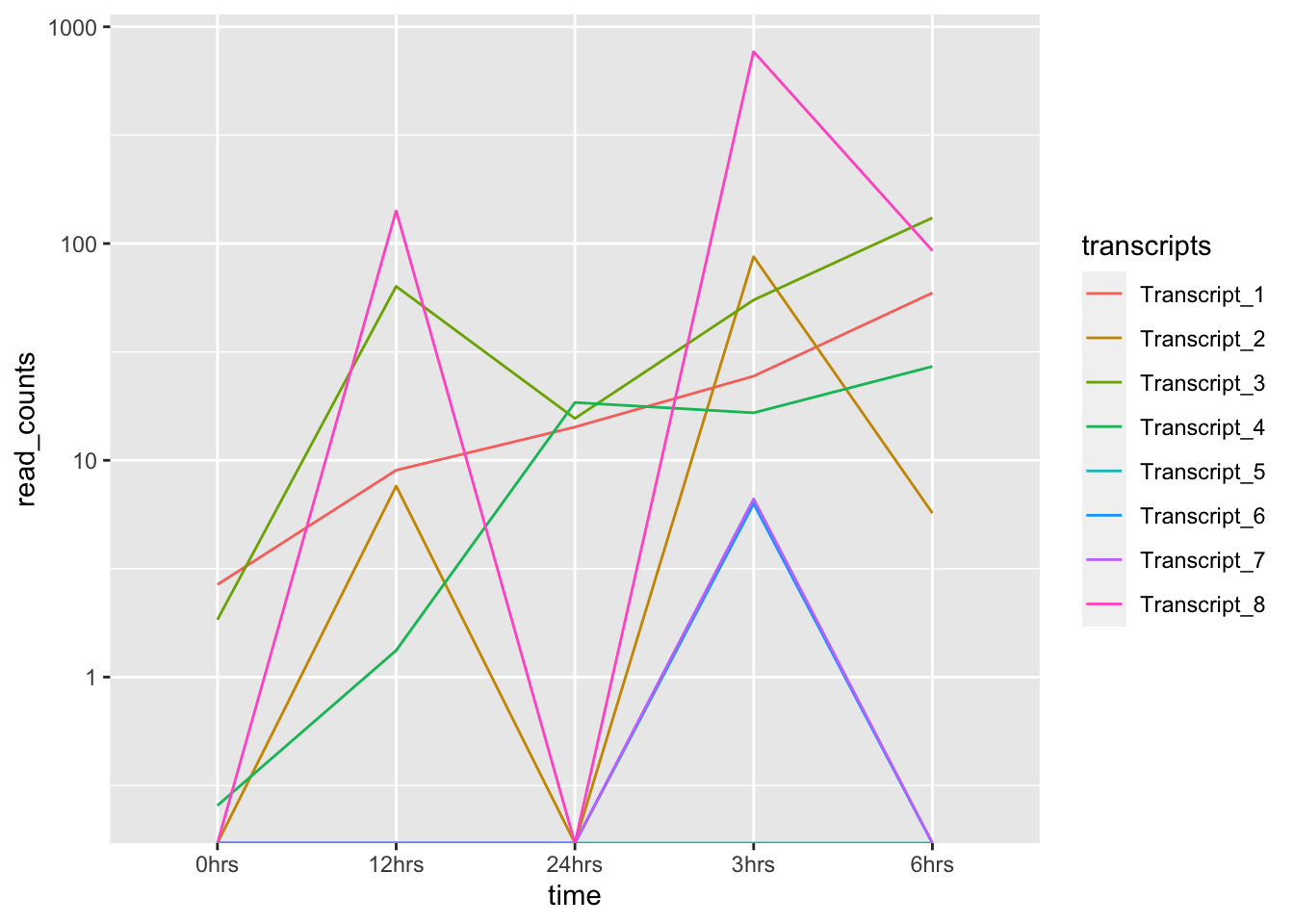
¡Warning message! Obtenemos un gráfico, pero algo no está bien…
¿A qué crees que se debe el mensaje de advertencia? ¿Qué otro aspecto del gráfico crees tú es necesario modificar?
- Solución:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts))
¡Truco! Transforma la columna “time” en un factor ordenado, para que ggplot respete el orden deseado de tu serie de tiempo:
tr_plot$time <- factor(tr_plot$time, levels=unique(tr_plot$time))
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts))
6.5 Faceting
Faceting es una propiedad de ggplot que permite dividir un gráfico en múltiples gráficos basado en un factor incluído en el set de datos.
- Continuando con el gráfico que hemos estado trabajando, usamos
facet_wrap()para hacer un gráfico por cada transcrito:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts)) +
facet_wrap(~transcripts)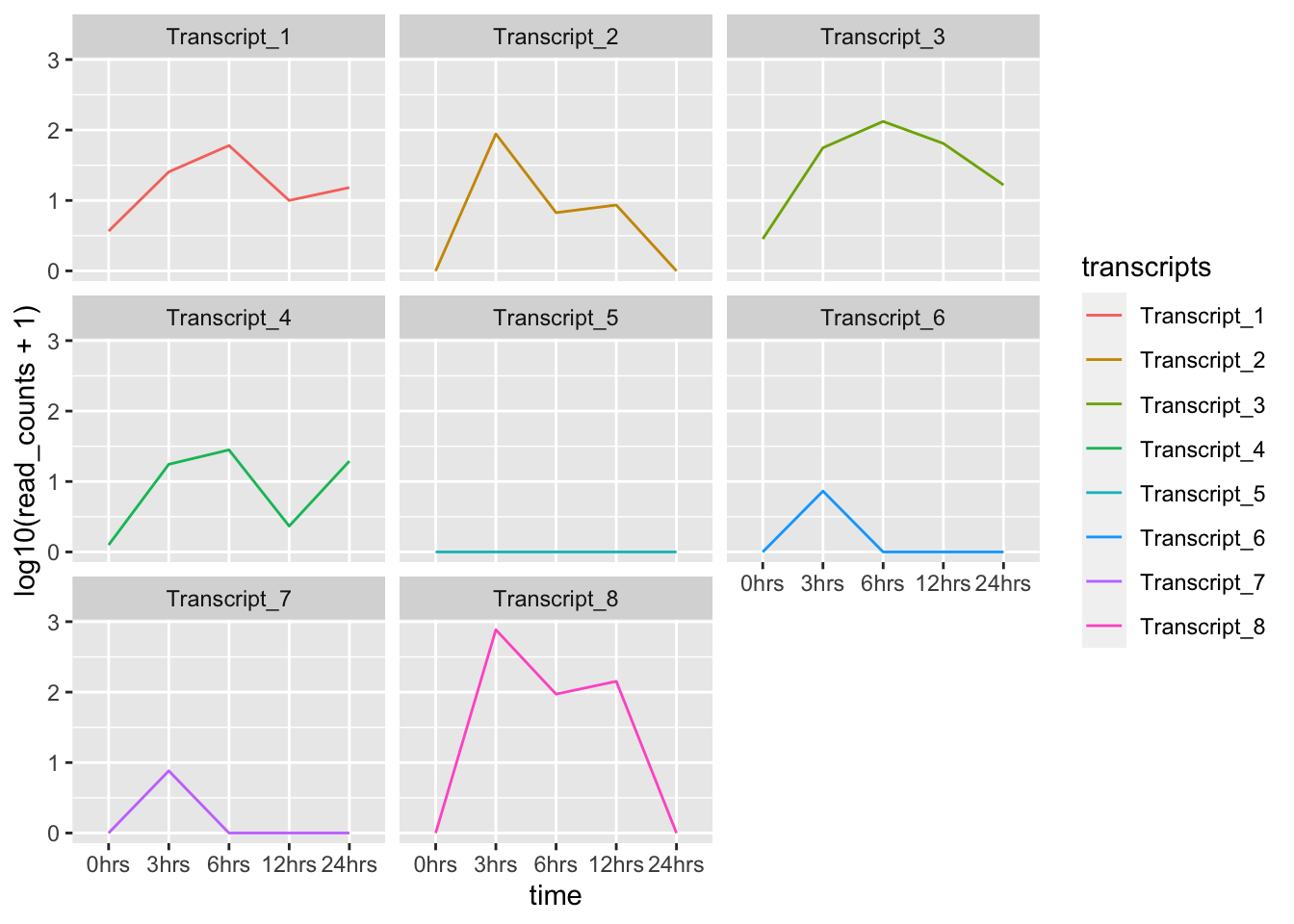
6.6 ggplot2 themes
Themes permite configurar la apariencia o características estéticas de los gráficos creados con ggplot. Puedes revisar la lista completa de themes disponibles aquí.
- Por ejemplo, gráficos con fondo blanco lucen mejor cuando son impresos. Podemos hacer que el fondo de las gráficas sea blanco usando la función
theme_bw(), también podemos remover la cuadrícula:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts)) +
facet_wrap(~ transcripts) +
theme_bw() +
theme(panel.grid = element_blank())
Mientras que, la función facet_wrap() organiza los gráficos en un número arbitrario de filas y columnas, también existe la función facet_grid que permite especificar como organizar los gráficos usando la siguiente nomenclatura: filas ~ columnas, también podemos usar . para indicar sólo una fila o columna (e.g., todas las gráficas en una columna: facet_grid(transcripts ~ .)).
6.7 Personalización
ggplot2 cuenta con varias funciones para personalizar gráficas, dale un vistazo al Cheat Sheet “Data Visualization” para pensar en formas de mejorar tus gráficos, también puedes inspirarte buscando ejemplos en internet, como aquí.
- Vamos a revisar un ejemplo de como personalizar el boxplot que estuvimos trabajando anteriormente:
ggplot(data = data, aes(x = geo_loc_name, y = observed, color = geo_loc_name)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5) +
coord_cartesian(ylim = c(10, 85)) +
scale_y_continuous(breaks = c(10,20,30,40,50,60,70,80)) +
theme(axis.text.x = element_text(face = "bold", angle = 45, hjust = 1, size = 10),
axis.title.x = element_blank(),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.position = "none") +
labs(y = "Observed number of taxa")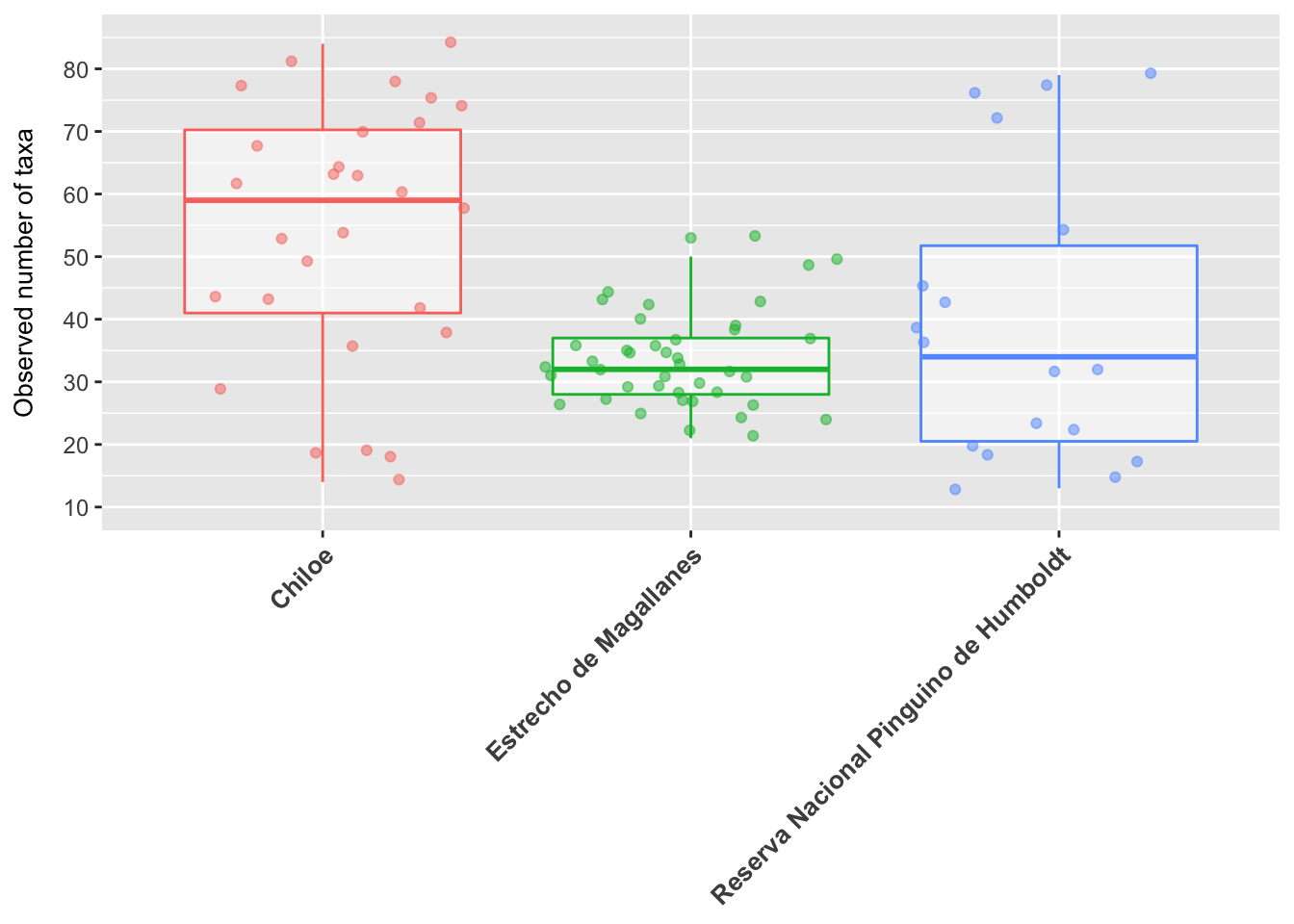
6.8 Organizar y exportar gráficos
Anteriormente usamos faceting para separar un gráfico en múltiples gráficos. Sin embargo, también podemos crear una figura con múltiples gráficos diferentes (i.e., diferentes variables, set de datos, tipo de gráfica). El paquete gridExtra nos permite combinar gráficos separados en una única figura, usando la función grid.arrange().
p1 <- ggplot(data = data, aes(x = geo_loc_name, y = observed, color = geo_loc_name)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5) +
coord_cartesian(ylim = c(10, 85)) +
scale_y_continuous(breaks = c(10,20,30,40,50,60,70,80)) +
theme(axis.text.x = element_text(face = "bold", angle = 45, hjust = 1, size = 10),
axis.title.x = element_blank(),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.position = "none") +
labs(y = "Observed number of taxa")
p2 <- ggplot(data = data, aes(x = shannon, y = evenness_camargo, color = species, shape = geo_loc_name)) +
geom_point(alpha = 0.7, size = 4) +
coord_cartesian(xlim = c(0.0,3.2), ylim = c(0.005,0.085)) +
theme(axis.text.x = element_text(size = 9),
axis.title.x = element_text(size = 10, margin = margin(t = 10, r = 0, b = 0, l = 0)),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.text = element_text(size = 10),
legend.title = element_blank(),
legend.position = c(.02, .98),
legend.justification = c("left", "top"),
legend.box.just = "left",
legend.margin = margin(2, 2, 2, 2)) +
labs(x = "Shannon", y = "Camargo evenness")
grid.arrange(p1, p2, ncol = 2, widths = c(1,2.5))
Después de crear tu gráfico o figura, puedes guardarlo en un archivo de formato de preferencia (e.g., png, pdf):
En RStudio, dirígete a la pestaña
Plotsy haz clic enExportpara guardar el gráfico que estés visualizando.También puedes usar la función
ggsave()para, no solo guardar tus gráficos, sino que también modificar la dimensión y resolución por medio de los argumentoswidth,heightydpi.