13 Introducción al análisis de diversidad
¿Qué entendemos por diversidad? Al menos podemos conceptualizar diversidad a dos niveles: diversidad genética o morfológica, y biodiversidad. En el estudio de comunidades, tomamos prestado el concepto de biodiversidad de ecología de comunidades donde estamos interesados en la riqueza de especies (número de especies diferentes en una comunidad o diversidad alfa), en las diferencias y similitudes entre comunidades (diversidad beta), y en algunos casos en la diversidad total de un región o paisaje ecológico (landscape; diversidad gama).

En la figura observamos la diversidad de aves en dos regiones, X e Y, y cuatro sitios, 1-4 (figura tomada de Community Ecology de Mittelbach). La diversidad alfa es mayor en los sitios 1 y 3 con 5 especies cada uno. La diversidad beta mide la cantidad de cambio o turnover de especies entre sitios. En la figura, la región Y tiene una diversidad beta mayor que la región X porque el cambio o turnover de especies entre el sitio 2 y el 4 es mayor que entre el sitio 1 y el 3 (que tienen las mismas 5 especies). La diversidad gama mide la diversidad total dentro de una región, por lo tanto en nuestro ejemplo la diversidad gama es mayor en la región Y porque contiene 6 especies en total versus la región X que tiene 5 especies.
13.1 Medidas de riqueza, uniformidad, dominancia, diversidad filogenética (diversidad alfa)
En el contexto metagenómico, medimos diversidad alfa usando una serie de medidas prestadas de ecología que nos permiten caracterizar una comunidad microbiana. phyloseq tiene una función muy útil que nos permite calcular y graficar hasta siete medidas, i .e., Observed (simplemente el número de taxa o riqueza), Chao1 (la riqueza ajustada por probabilidad de no observar especies), ACE (riqueza que toma en cuenta la abundancia relativa), Shannon (abundancia relativa de taxa), Simpson (1 - la probabilidad de que observemos aleatoriamente dos bacterias en una comunidad y que pertenezcan a diferentes especies ), Inverse Simpson ( 1 / Simpson), y Fisher (riqueza tomando en cuenta abundancia).
- En
phyloseqsimplemente llamamos la funciónplot_richnessy podemos visualizar las medidas de diversidad.
plot_richness(psd5, color = "species", x = "species", measures = c("Observed", "Chao1", "Shannon")) + geom_boxplot(aes(fill = species), alpha=.7) + scale_color_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f")) + scale_fill_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f"))
En el ejemplo, solo graficamos Observed, Chao1 y Shannon usando el argumento measures = c("Observed", "Chao1", "Shannon"). Si quisieramos obtener todas las medidas simplemente eliminamos este argumento y por defecto phyloseq graficará todo.
¿Hay un efecto significativo de la diversidad alfa según especie de ballena? Eso lo podríamos probar rápidamente con un análisis de varianza (ANOVA). Para este ejemplo utilicemos otra medida de diversidad, una que phyloseq no incorpora. Faith’s Phylogenetic Diversity es un índice introducido por Daniel Faith en 1992 que no solo considera número de especies sino que también considera qué tanto se parecen estas especies filogenéticamente. Esto es muy relevante porque nos entrega una medida rápida para evaluar prioridades de conservación de ecosistemas, o si se trata de comunidades microbianas, donde tenemos mayor probabilidad de encontrar funciones génicas novedosas.
# Guardamos un dataframe con las medidas de diversidad alfa
alpha_pd <- estimate_pd(psd5)
# Combinamos la metadata con alpha.diversity
data <- cbind(sample_data(psd5), alpha_pd)
# Y calculamos un ANOVA
psd5.anova <- aov(PD ~ species, data)
# install.packages("xtable")
library(xtable)
psd5.anova.table <- xtable(psd5.anova)
kable(psd5.anova.table, caption = "Tabla ANOVA", digits = 5, format = "markdown")| Df | Sum Sq | Mean Sq | F value | Pr(>F) | |
|---|---|---|---|---|---|
| species | 2 | 40.46002 | 20.23001 | 5.21047 | 0.00741 |
| Residuals | 82 | 318.37078 | 3.88257 | NA | NA |
- El paquete
microbiomeofrece otras herramientas para evaluar diversidad que son accesibles fácilmente a través de su funciónglobal.
tab <- global(psd5, index = "all")
head(tab) %>%
kable(format = "html", col.names = colnames(tab), digits = 2) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "300px")| observed | chao1 | diversity_inverse_simpson | diversity_gini_simpson | diversity_shannon | diversity_fisher | diversity_coverage | evenness_camargo | evenness_pielou | evenness_simpson | evenness_evar | evenness_bulla | dominance_dbp | dominance_dmn | dominance_absolute | dominance_relative | dominance_simpson | dominance_core_abundance | dominance_gini | rarity_log_modulo_skewness | rarity_low_abundance | rarity_rare_abundance | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SRR6442697 | 31 | 49.00 | 6.95 | 0.86 | 2.18 | 4.10 | 3 | 0.76 | 0.64 | 0.22 | 0.10 | 0.30 | 0.27 | 0.40 | 2099 | 0.27 | 0.14 | 0.26 | 0.95 | 2.06 | 0.01 | 0.13 |
| SRR6442698 | 38 | 41.50 | 3.67 | 0.73 | 1.91 | 5.78 | 2 | 0.29 | 0.53 | 0.10 | 0.14 | 0.28 | 0.49 | 0.62 | 2015 | 0.49 | 0.27 | 0.67 | 0.96 | 2.06 | 0.02 | 0.10 |
| SRR6442699 | 25 | 37.25 | 6.46 | 0.85 | 2.04 | 3.12 | 3 | 0.57 | 0.63 | 0.26 | 0.07 | 0.31 | 0.26 | 0.41 | 2461 | 0.26 | 0.15 | 0.47 | 0.96 | 2.04 | 0.00 | 0.14 |
| SRR6442700 | 53 | 63.00 | 7.71 | 0.87 | 2.41 | 6.10 | 3 | 0.18 | 0.61 | 0.15 | 0.08 | 0.27 | 0.23 | 0.42 | 8300 | 0.23 | 0.13 | 0.61 | 0.94 | 2.05 | 0.01 | 0.01 |
| SRR6442701 | 48 | 68.25 | 4.52 | 0.78 | 2.04 | 5.95 | 2 | 0.28 | 0.53 | 0.09 | 0.10 | 0.24 | 0.41 | 0.54 | 7742 | 0.41 | 0.22 | 0.23 | 0.95 | 2.05 | 0.01 | 0.03 |
| SRR6442702 | 42 | 82.50 | 4.87 | 0.79 | 1.76 | 4.35 | 3 | 0.12 | 0.47 | 0.12 | 0.07 | 0.15 | 0.28 | 0.49 | 19137 | 0.28 | 0.21 | 0.46 | 0.97 | 2.05 | 0.01 | 0.00 |
La función global nos da 26 medidas de diversidad que nos ayudan a entender la estructura de las comunidades microbianas. En general, estas medidas se dividen en riqueza, diversidad, dominancia, rareza, cobertura y uniformidad.
- El paquete
microbiomeofrece funciones para calcular cada uno de estos aspectos de las comunidades microbianas.
# Riqueza
tab <- richness(psd5)
# Dominancia
tab <- dominance(psd5, index = "all")
# Rareza
tab <- rarity(psd5, index = "all")
# Cobertura
tab <- coverage(psd5, threshold = 0.5)
# Desigualdad
tab <- inequality(psd5)
# Uniformidad
tab <- evenness(psd5, "all")- Veamos un ejemplo concreto estimando diversidad, graficando los resultados y calculando significancia estadística. Para esto usamos el paquete
ggpubrque genera “publication-ready plots”, algo que siempre es deseable (ejecutalibrary(ggpubr)).
# Generamos un objeto `phyloseq` sin taxa que sume 0 reads
psd5.2 <- prune_taxa(taxa_sums(psd5) > 0, psd5)
# Calculamos los índices de diversidad
tab <- diversities(psd5.2, index = "all")
# Y finalmente visualizamos la tabla de resultados
head(tab) %>%
kable(format = "html", col.names = colnames(tab), digits = 2) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "310px")| observed | chao1 | diversity_inverse_simpson | diversity_gini_simpson | diversity_shannon | diversity_fisher | diversity_coverage | evenness_camargo | evenness_pielou | evenness_simpson | evenness_evar | evenness_bulla | dominance_dbp | dominance_dmn | dominance_absolute | dominance_relative | dominance_simpson | dominance_core_abundance | dominance_gini | rarity_log_modulo_skewness | rarity_low_abundance | rarity_rare_abundance | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SRR6442697 | 31 | 49.00 | 6.95 | 0.86 | 2.18 | 4.10 | 3 | 0.76 | 0.64 | 0.22 | 0.10 | 0.30 | 0.27 | 0.40 | 2099 | 0.27 | 0.14 | 0.26 | 0.95 | 2.06 | 0.01 | 0.13 |
| SRR6442698 | 38 | 41.50 | 3.67 | 0.73 | 1.91 | 5.78 | 2 | 0.29 | 0.53 | 0.10 | 0.14 | 0.28 | 0.49 | 0.62 | 2015 | 0.49 | 0.27 | 0.67 | 0.96 | 2.06 | 0.02 | 0.10 |
| SRR6442699 | 25 | 37.25 | 6.46 | 0.85 | 2.04 | 3.12 | 3 | 0.57 | 0.63 | 0.26 | 0.07 | 0.31 | 0.26 | 0.41 | 2461 | 0.26 | 0.15 | 0.47 | 0.96 | 2.04 | 0.00 | 0.14 |
| SRR6442700 | 53 | 63.00 | 7.71 | 0.87 | 2.41 | 6.10 | 3 | 0.18 | 0.61 | 0.15 | 0.08 | 0.27 | 0.23 | 0.42 | 8300 | 0.23 | 0.13 | 0.61 | 0.94 | 2.05 | 0.01 | 0.01 |
| SRR6442701 | 48 | 68.25 | 4.52 | 0.78 | 2.04 | 5.95 | 2 | 0.28 | 0.53 | 0.09 | 0.10 | 0.24 | 0.41 | 0.54 | 7742 | 0.41 | 0.22 | 0.23 | 0.95 | 2.05 | 0.01 | 0.03 |
| SRR6442702 | 42 | 82.50 | 4.87 | 0.79 | 1.76 | 4.35 | 3 | 0.12 | 0.47 | 0.12 | 0.07 | 0.15 | 0.28 | 0.49 | 19137 | 0.28 | 0.21 | 0.46 | 0.97 | 2.05 | 0.01 | 0.00 |
- Ahora necesitamos extraer la metadata de nuestro objeto
phyloseq.
psd5.2.meta <- meta(psd5.2)
head(psd5.2.meta) %>%
kable(format = "html", col.names = colnames(psd5.2.meta), digits = 2) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "500px")| sample_ID | bioproject_accession | study | biosample_accession | experiment | run | SRA_Sample | geo_loc_name | collection_date | sample_type | species | common_name | AvgSpotLen | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SRR6442697 | EMA4 | PRJNA428495 | SRP128093 | SAMN08292292 | SRX3533985 | SRR6442697 | SRS2809259 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442698 | EMA3 | PRJNA428495 | SRP128093 | SAMN08292291 | SRX3533984 | SRR6442698 | SRS2809258 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442699 | EMA2 | PRJNA428495 | SRP128093 | SAMN08292284 | SRX3533983 | SRR6442699 | SRS2809257 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442700 | EMA19 | PRJNA428495 | SRP128093 | SAMN08292283 | SRX3533982 | SRR6442700 | SRS2809256 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442701 | EMA21 | PRJNA428495 | SRP128093 | SAMN08292286 | SRX3533981 | SRR6442701 | SRS2809255 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442702 | EMA20 | PRJNA428495 | SRP128093 | SAMN08292285 | SRX3533980 | SRR6442702 | SRS2809254 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
- Luego agregamos la tabla de diversidad a la metadata.
psd5.2.meta$Shannon <- tab$diversity_shannon
psd5.2.meta$InverseSimpson <- tab$diversity_inverse_simpson- En este ejercicio nos interesa comparar la diversidad entre especies de ballenas. Recordemos que tenemos datos para tres especies de ballenas: azul, fin y jorobada. Necesitamos crear una lista de comparasiones de a pares para poder visualizar y calcular significancia estadística de manera simultánea.
# Obtenemos las variables desde nuestro objeto `phyloseq`
spps <- levels(psd5.2.meta$species)
# Creamos una lista de lo que queremos comparar
pares.spps <- combn(seq_along(spps), 2, simplify = FALSE, FUN = function(i)spps[i])
# Imprimimos en pantalla el resultado
print(pares.spps)## [[1]]
## [1] "Balaenoptera musculus" "Balaenoptera physalus"
##
## [[2]]
## [1] "Balaenoptera musculus" "Megaptera novaeangliae"
##
## [[3]]
## [1] "Balaenoptera physalus" "Megaptera novaeangliae"- Con la función
ggviolinpodemos generar un gráfico de violín en un solo paso de la siguiente forma.
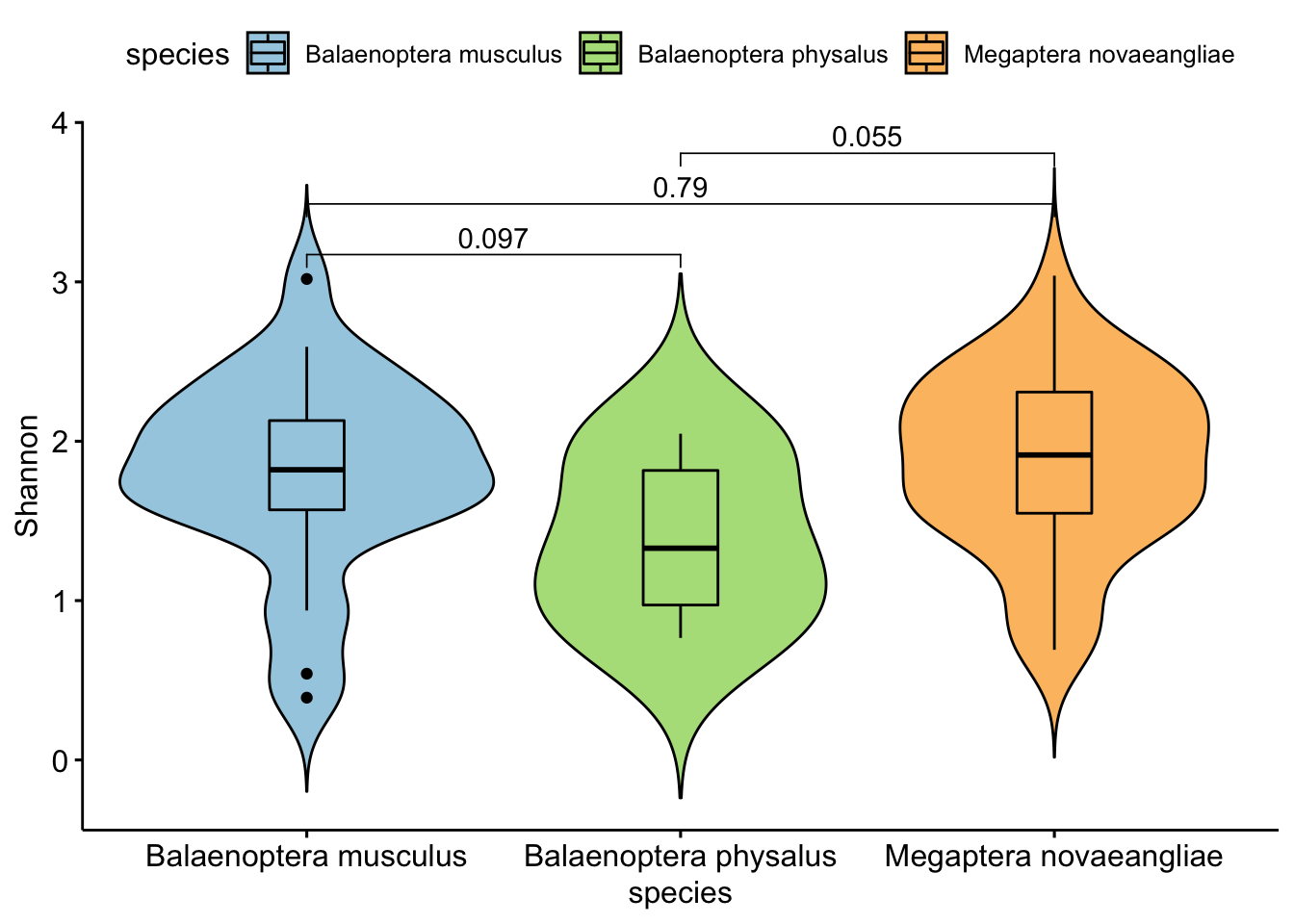
p1 <- ggviolin(psd5.2.meta, x = "species", y = "Shannon",
add = "boxplot", fill = "species", palette = c("#a6cee3", "#b2df8a", "#fdbf6f"))
print(p1)
- Ahora necesitamos evaluar la significancia estadística entre los estimados de diversidad de las muestras de ballenas. De nuevo, en una línea, tenemos nuestra figura lista para el artículo.
13.2 Diversidad beta y escalamiento multidimensional (Bray-Curtis, UniFrac, t-SNE)
En cuanto a diversidad beta podemos calcular similitud global a través de todas las muestras de interés o también podemos cuantificar la divergencia de un grupo y compararla con la divergencia de otro.
- Veamos este último caso primero.
# Calculamos las divergencias para ballena azul y jorobada
div.azul <- divergence(subset_samples(psd5, species == "Balaenoptera musculus"), apply(abundances(subset_samples(psd5, species == "Balaenoptera musculus")), 1, median))
div.joro <- divergence(subset_samples(psd5, species == "Megaptera novaeangliae"), apply(abundances(subset_samples(psd5, species == "Megaptera novaeangliae")), 1, median))
# transformamos el resultado anterior en _dataframes_
data.frame(div.azul) -> df.div.azul
data.frame(div.joro) -> df.div.joro
# Gather
library(tidyverse)
df.div.azul <- gather(df.div.azul, sample, divergence)
df.div.joro <- gather(df.div.joro, sample, divergence)
# Agregamos columnas a nuestros _dataframes_
mutate(df.div.azul, species = "Blue Whale") -> df.div.azul
mutate(df.div.joro, species = "Humpback Whale") -> df.div.joro
# Cambiamos los nombres de las columans de manera que sean iguales an ambos _dataframes_
colnames(df.div.azul) <- c("samples", "divergence", "species")
colnames(df.div.joro) <- c("samples", "divergence", "species")
# Los combinamos en un _dataframe_
rbind(df.div.azul, df.div.joro) -> div.boxplot
# Y finalmente graficamos y realizamos una comparación estadística
p2 <- ggboxplot(data = div.boxplot, x = "species", y = "divergence", fill = "species", palette = c("#a6cee3", "#fdbf6f"))
p2 + stat_compare_means(comparisons = list(c("Blue Whale", "Humpback Whale")))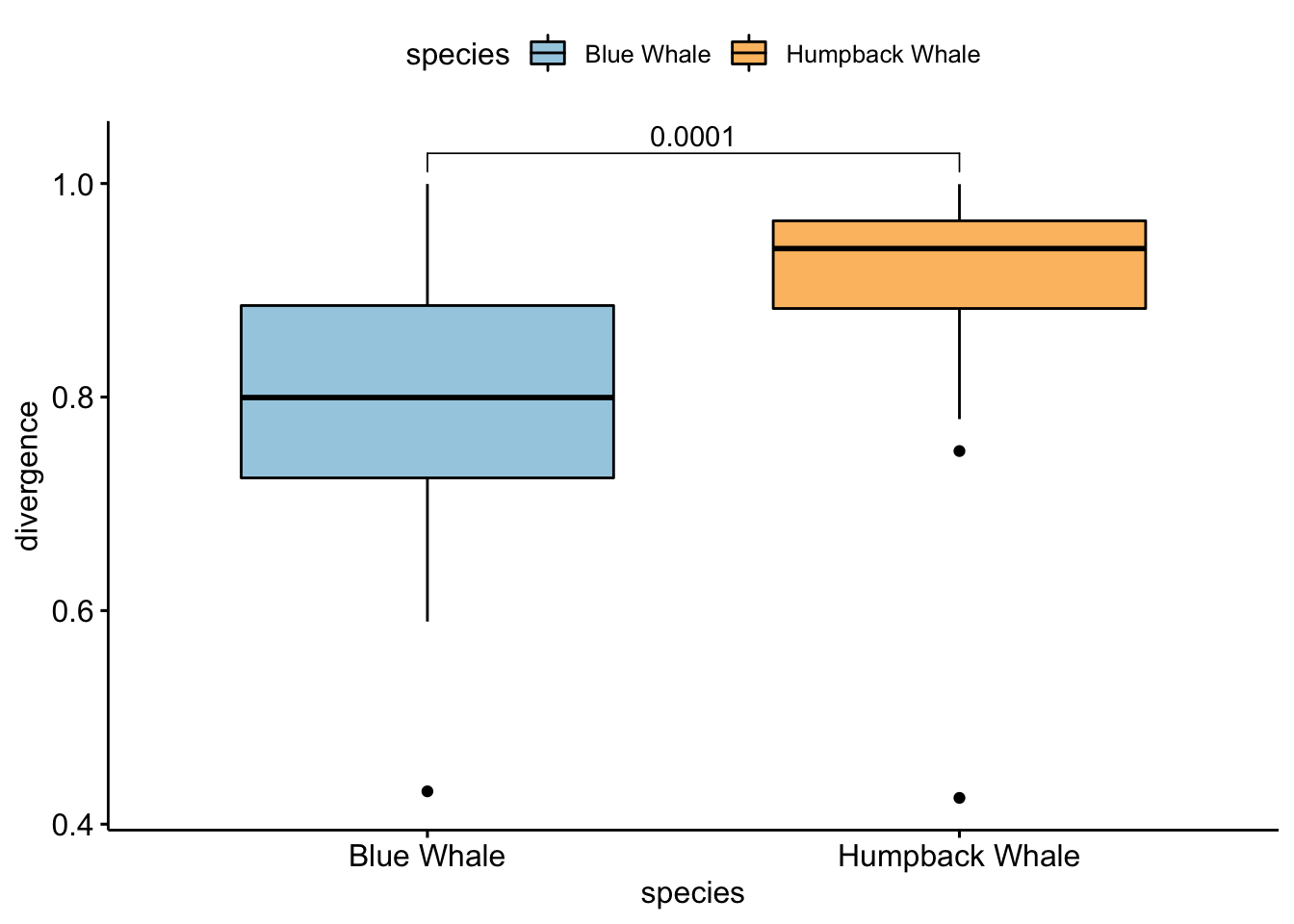
Existen diferentes medidas de similitud (o disimilitud) disponibles que nos permiten entender las relaciones entre nuestras muestras. En general todas producen matrices de distancia comparables. El paquete phyloseq ofrece un gran número de medidas de distancia. Las más populares son UniFrac y Weighted UniFrac (medidas que consideran filogenia) y otras independientes de filogenia como: Jaccard, Manhattan, Euclidian, Bray-Curtis, Canberra, etc. Por otra parte, la matriz de distancia resultante no se usa en aislación sino que en conjunto con algún método de ordinación o escalamiento multidimensional (ordination). De nuevo, phyloseq ofrece un gran número de métodos entre los cuales se encuentran: detrended y canonical correspondence analysis, Double Principle Coordinate Analysis, Non-metric MultiDimenstional Scaling, y MDS/PCoA.
- Probemos entonces hacer un análisis tipo PCoA con una matriz de distancia que considera las relaciones filogenéticas y otra que no.
psd5.mds.unifrac <- ordinate(psd5, method = "MDS", distance = "unifrac")
evals <- psd5.mds.unifrac$values$Eigenvalues
pord1 <- plot_ordination(psd5, psd5.mds.unifrac, color = "geo_loc_name") +
labs(col = "geo_loc_name") +
coord_fixed(sqrt(evals[2] / evals[1]))
psd5.mds.bray <- ordinate(psd5, method = "MDS", distance = "bray")
evals <- psd5.mds.bray$values$Eigenvalues
pord2 <- plot_ordination(psd5, psd5.mds.bray, color = "geo_loc_name") +
labs(col = "geo_loc_name") +
coord_fixed(sqrt(evals[2] / evals[1]))
grid.arrange(pord1, pord2)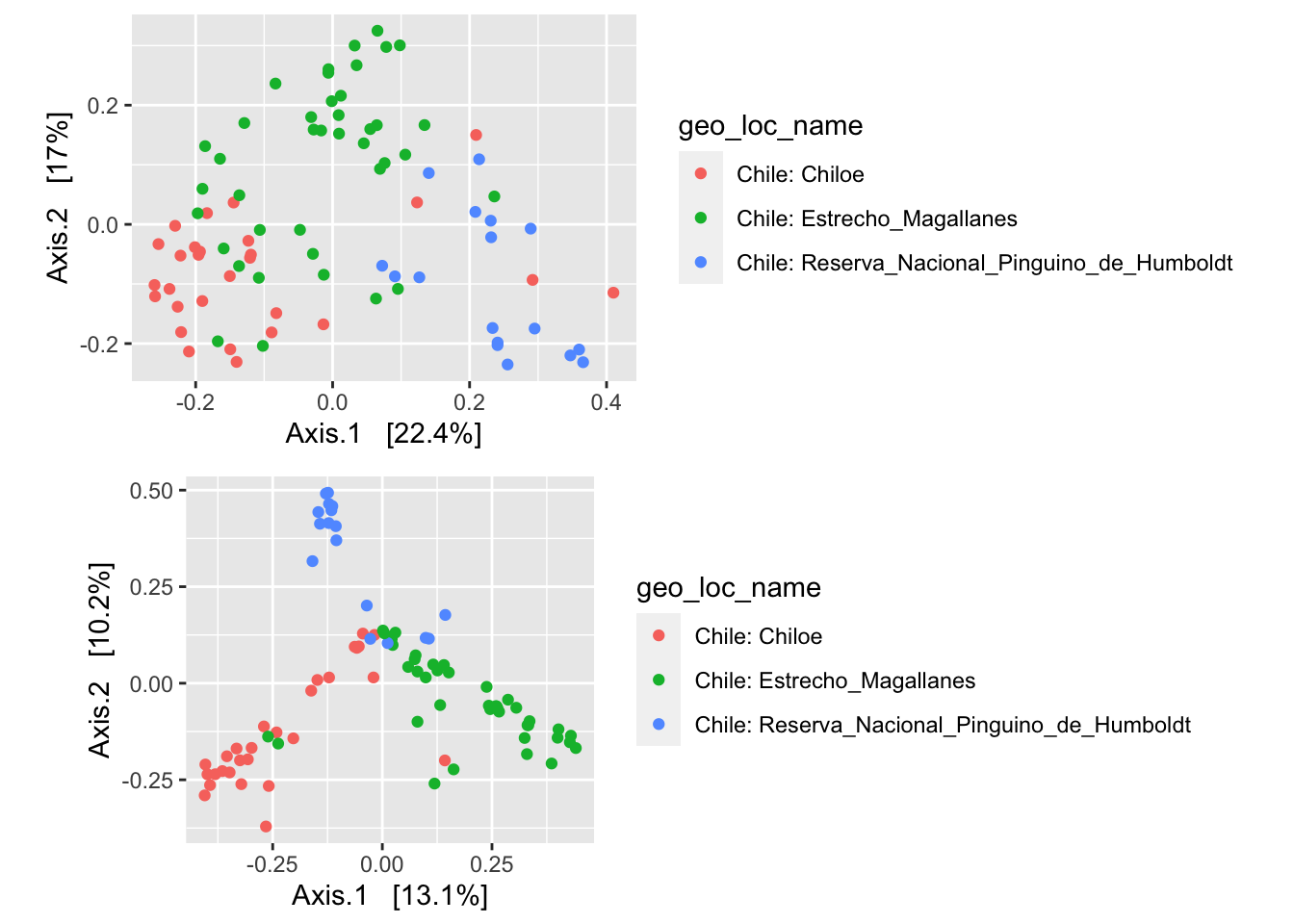
Nota que los gráficos de dispersión donde se visualiza este tipo de análisis están escalados según la cantidad de variación que los ejes explican. En general lo que estos métodos pretenden hacer es tratar de encontrar el menor número de vectores matemáticos que maximicen la separación entre las muestras (puntos en el gráfico). Esto nace de la imposibilidad de graficar eficientemente datos multidimencionales. Los datos que estamos analizando ciertamente son multidimensionales en el sentido que tenemos más de 100 taxa que varían simultáneamente en cada una de las 90+ muestras que tenemos. Volviendo a los ejes, estos no suman 100% porque hay otros ejes que no estamos usando para graficar y que contribuyen con el resto de la variación. Al graficarlos de manera simétrica distorsionaríamos las relaciones entre los puntos, especialmente si estamos comparando dos o más gráficos.
En específico para comunidades microbianas, el método de doble análisis de coordenadas principales o (DPCoA) es muy apropiado porque analiza conjuntamente dos tipos de datos: una tabla de disimilitud que representa diferencias entre especies y una matriz de abundancia que representa la distribución de especies entre las comunidades. El resultado final es un ensamble del espacio multidimensional que correlacionan las especies con las comunidades. El método fue publicado en 2004.
- Veamos un ejemplo con nuestros datos.
psd5.dpcoa.unifrac <- ordinate(psd5, method = "DPCoA", distance = "dpcoa")
evals <- psd5.dpcoa.unifrac$eig
pord3 <- plot_ordination(psd5, psd5.dpcoa.unifrac, color = "species", shape = "geo_loc_name") +
labs(col = "Species") +
coord_fixed(sqrt(evals[2] / evals[1])) +
scale_color_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f")) +
scale_fill_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f")) +
geom_point(size=4)
pord3
- Ahora exploremos escalamiento multidimensional usando un método reciente conocido como t-SNE o t-Distributed Stochastic Neighbor Embedding. t-SNE difiere de otros métodos en que hace énfasis en las distancias locales en vez de las distancias globales, de esta forma generando una mayor resolución o separación entre los puntos o muestras.
library(tsnemicrobiota)
tsne_res <- tsne_phyloseq(psd5, distance= "dpcoa", perplexity = 8, verbose=0, rng_seed = 3901)
# Graficamos
pord4 <- plot_tsne_phyloseq(psd5, tsne_res, color = "species", shape = "geo_loc_name") +
geom_point(size=4) +
scale_color_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f")) +
scale_fill_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f"))
grid.arrange(pord3, pord4)
Ambos gráficos usan distintos métodos pero la misma medida de distancia. Los resultados son similares aunque las agrupaciones de puntos o muestras ocupan distinto espacio en el gráfico.
- Otro uso de estas medidas es a través de la visualización de la densidad de las muestras en el espacio.
method <- "tsne"
trans <- "hellinger"
distance <- "euclidean"
# Matriz de distancia
psd5 <- microbiome::transform(psd5, trans)
# Calculamos similitud entre muestras
dm <- vegdist(otu_table(psd5), distance)
# Corremos t-SNE
tsne_out <- Rtsne(dm, dims = 2, perplexity = 8)
proj <- tsne_out$Y
rownames(proj) <- rownames(otu_table(psd5))
data.frame(proj) -> proj
proj$species <- sample_data(psd5)[,11]
pland <- plot_landscape(proj[,1:2], legend = T, size = 4)
print(pland)
13.3 Análisis de abundancias y visualizaciones
13.3.1 Gráfico de barras apiladas
Una manera muy eficiente de obtener una visión general de la composición de las muestras es a través de un gráfico de barras apiladas. Existe una variadad de funciones que pueden hacer esto, sin embargo vamos a usar el paquete creado por un ex-miembro del laboratorio ya que tiene la ventaja de poder agrupar por hierarchical clustering las muestras entre otras ventajas.
# Necesitamos obtener las taxa más abundantes, en este caso el top 15
top15 <- get_top_taxa(physeq_obj = psd5, n = 15, relative = T,
discard_other = T, other_label = "Other")
# Ya que no todas las taxa fueron clasificadas a nivel de especie, generamos etiquetas compuestas de distintos rangos taxonómicos para el gráfico
top15 <- name_taxa(top15, label = "", species = F, other_label = "Other")
# Finalmente graficamos
fantaxtic_bar(top15, color_by = "Family", label_by = "Genus", facet_by = NULL, grid_by = NULL, other_color = "Grey") -> ptop15## Level N.color.shades Central.color
## 1 Cardiobacteriaceae 1 #6495ED
## 2 Flavobacteriaceae 1 #EDBC64
## 3 Moraxellaceae 5 #9A64ED
## 4 Gammaproteobacteria (Class) 2 #B7ED64
## 5 Xanthomonadaceae 1 #ED64DA
## 6 Enterobacteriaceae 2 #64ED77
## 7 Pseudomonadaceae 2 #ED6473
## 8 Burkholderiaceae 1 #64EDDE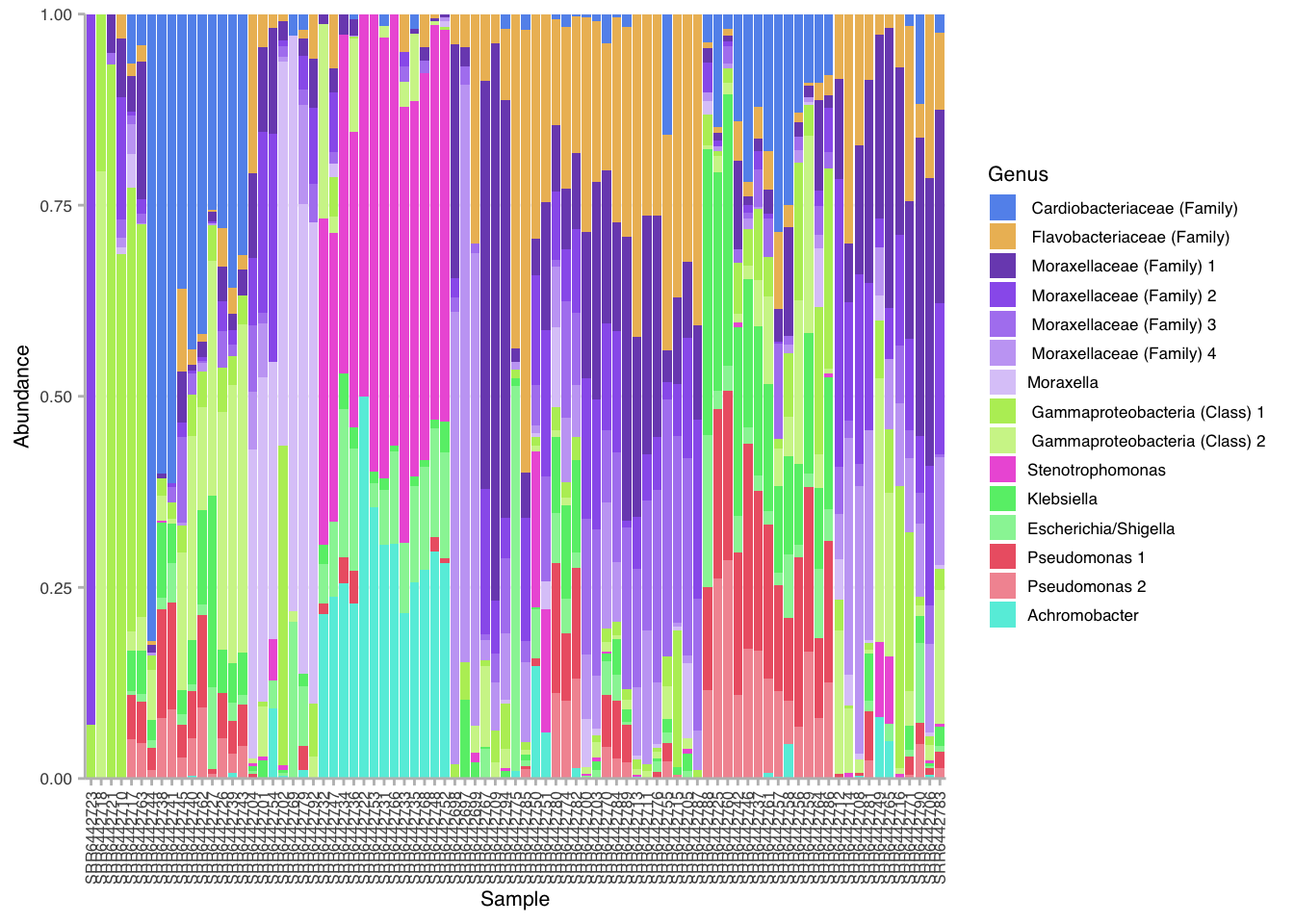
Podemos ver que existen patrones relativamente claros en el gráfico solamente a partir de los colores. Aunque tenemos el nombre de las muestras bajo cada barra, sería mejor poder parcelar este gráfico de manera que quede claro qué muestras corresponden a qué especie de ballena.
- La función
fantaxtic_barofrece estas posibilidades a través de los argumentosfacet_byygrid_by. Grafiquemos de nuevo.
fantaxtic_bar(top15, color_by = "Family", label_by = "Genus", facet_by = "species", grid_by = NULL, other_color = "Grey") -> ptop15.2## Level N.color.shades Central.color
## 1 Cardiobacteriaceae 1 #6495ED
## 2 Flavobacteriaceae 1 #EDBC64
## 3 Moraxellaceae 5 #9A64ED
## 4 Gammaproteobacteria (Class) 2 #B7ED64
## 5 Xanthomonadaceae 1 #ED64DA
## 6 Enterobacteriaceae 2 #64ED77
## 7 Pseudomonadaceae 2 #ED6473
## 8 Burkholderiaceae 1 #64EDDE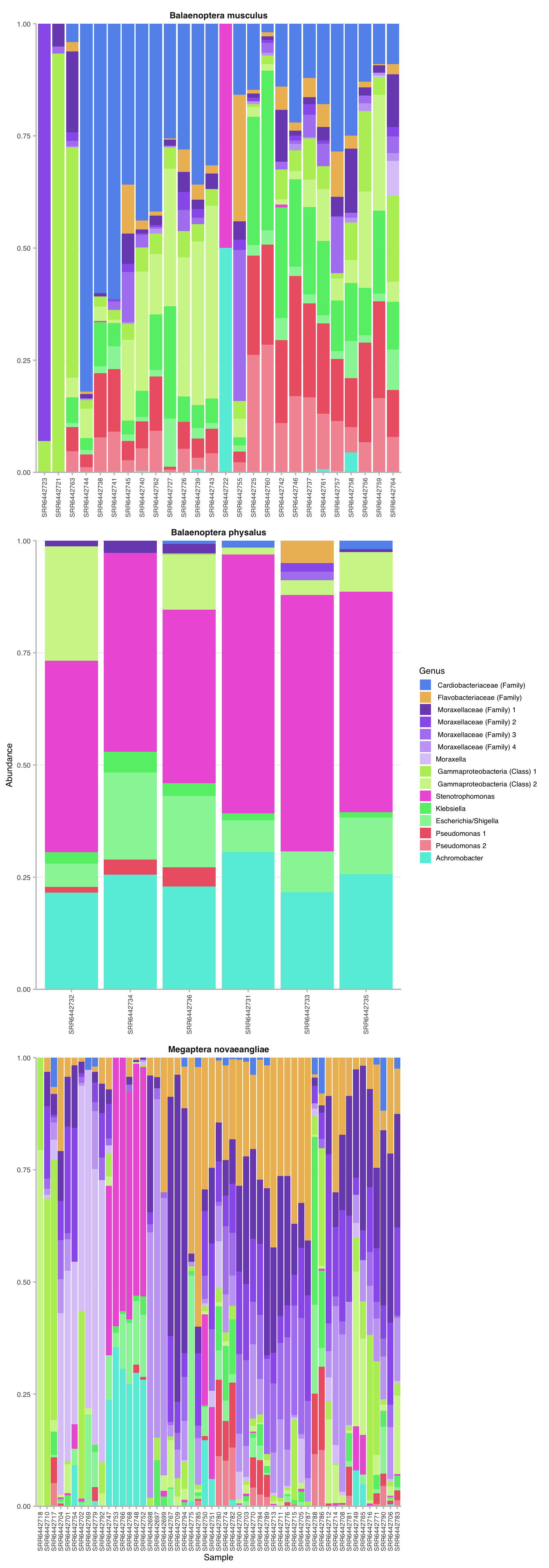
Ahora queda más claro y se puede observar que las distintas especies de ballena tienen un patrón similar entre sí que es diferente entre las otras, con algunas excepciones. Por ejemplo, para ballena jorobada podemos ver un conjunto de muestras que no se parecen al resto. Usando las herramientas ya aprendidas, ¿A qué corresponden esas muestras?
13.3.2 Diferentes visualizaciones de abundancias
Veamos ahora otras herramientas que nos permiten examinar la composición de estas comunidades microbianas. El paquete ampvis2, desarrollado por Mads Albertsen y Kasper Skytte Andersen, nos permite hacer precisamente esto. Primero transformemos el objeto phyloseq con el cual hemos estado trabajando en un objeto ampvis2.
library(ampvis2)
# Necesitamos extraer la tabla de read counts y la tabla de taxonomía del objeto psd5
# Generamos una copia para no sobreescribir psd5
obj <- psd5
# Cambiamos la orientación de la otu_table
t(otu_table(obj)) -> otu_table(obj)
# Extraemos las tablas
otutable <- data.frame(OTU = rownames(phyloseq::otu_table(obj)@.Data),
phyloseq::otu_table(obj)@.Data,
phyloseq::tax_table(obj)@.Data,
check.names = FALSE
)
# Extraemos la metadada
metadata <- data.frame(phyloseq::sample_data(obj),
check.names = FALSE
)
# ampvis2 requiere que 1) los rangos taxonómicos sean siete y vayan de Kingdom a Species y 2) la primera columna de la metadata sea el identificador de cada muestra
# Entonces duplicamos la columna Género y le cambiamos el nombre a Especie
otutable$Species = otutable$Genus
# Reordenamos la metadata
metadata <- metadata[,c("run","sample_ID","bioproject_accession","study","biosample_accession","experiment","SRA_Sample","geo_loc_name","collection_date","sample_type","species","common_name","AvgSpotLen")]
# finalmente generamos el objeto ampvis
av2 <- amp_load(otutable, metadata)- Ahora echemos un vistazo a la estructura de las comunidades utilizando “rank abundance curves”.

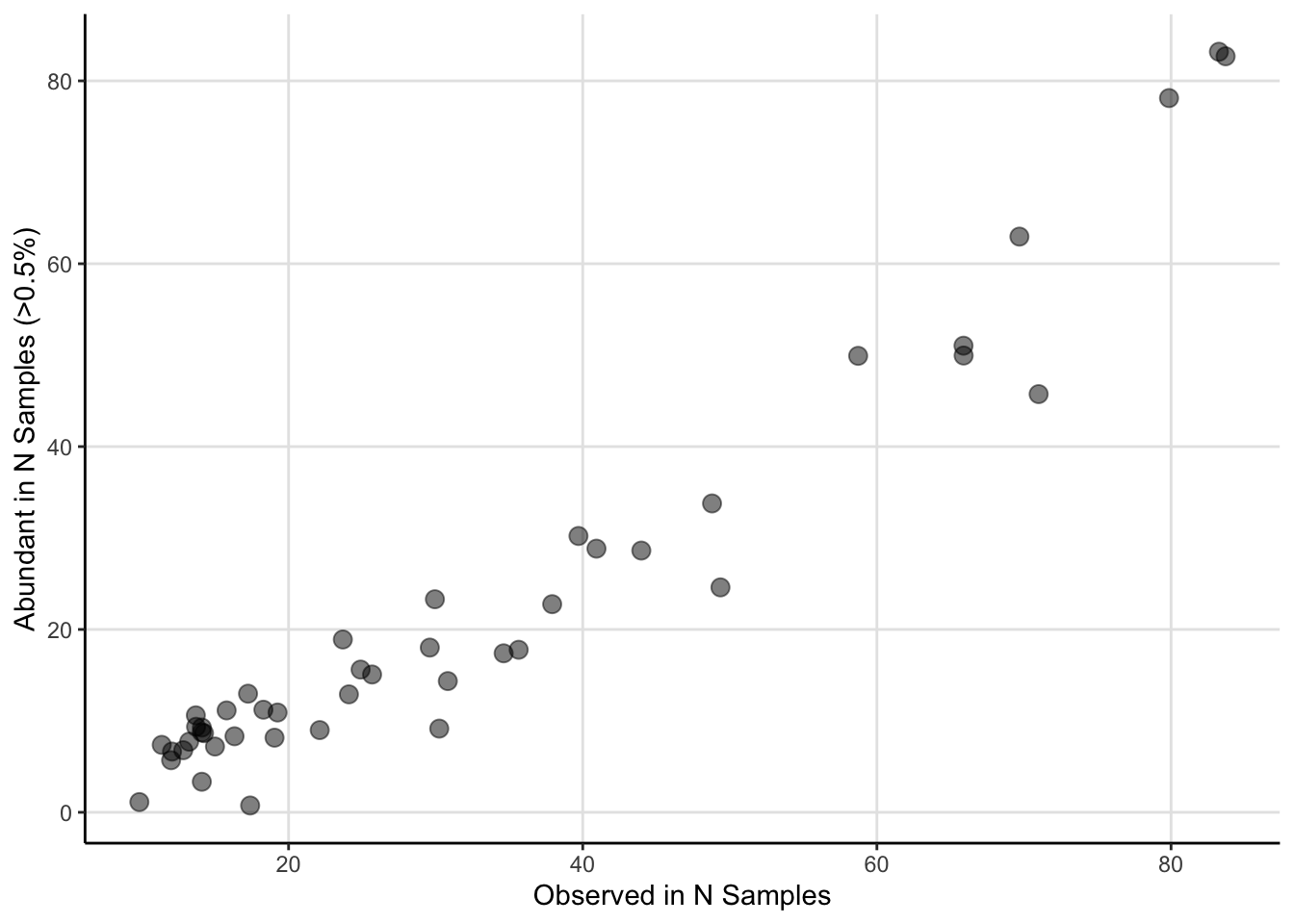
El gráfico nos muestra que en la medida que vamos sumando las taxa de mayor a menor abundancia (Rank Abundance) la abundancia de reads cumulativa va aumentando. Lo importante de observar es la forma de la curva. Una curva que sube rápidamente nos indica que las comunidades están dominadas por unas cuantas taxa. En cambio, lo que observamos en nuestros datos es que las taxa más abundantes solamente dan cuenta de aproximadamente el 25% de la abundancia cumulativa de reads.
- Veamos ahora qué taxa corresponde a ese 25%. Para esto podemos usar la función
amp_heatmap.
amp_heatmap(av2,
group_by = "species",
facet_by = "geo_loc_name",
plot_values = TRUE,
tax_show = 20,
tax_aggregate = "Genus",
tax_add = "Phylum",
plot_colorscale = "sqrt",
plot_legendbreaks = c(1, 5, 10)
)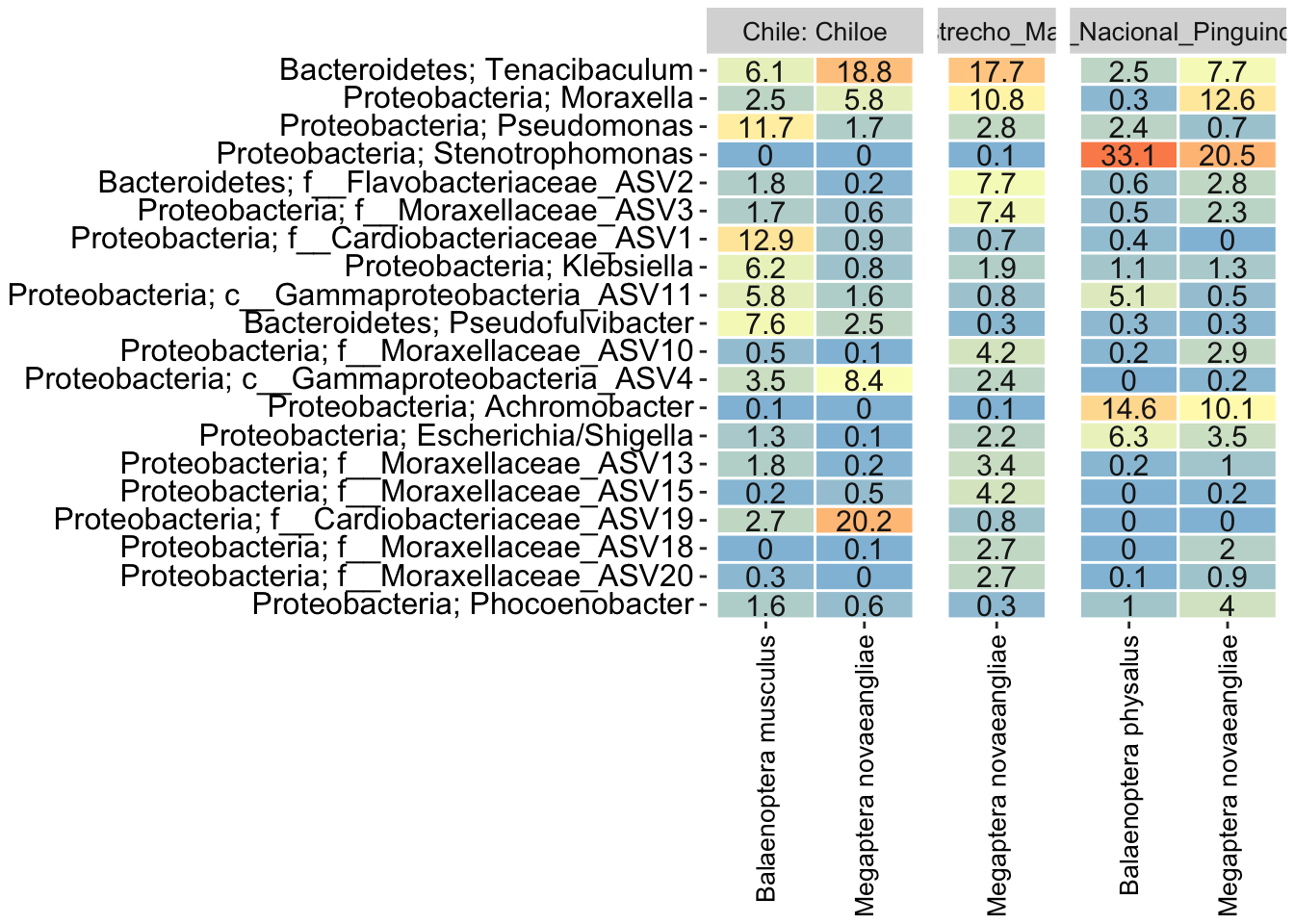
Tenacibaculum parece ser el género más abundante para todas las muestras en todas los sitios de muestreo con excepción de Balaenoptera physalus que está dominada por Stenotrphomonas. Moraxella y distintas variantes de Cardiobacteriaceae de género no conocido. Justamente estos resultados se ajustan a lo conocido para cetáceos y otros mamíferos marinos.
- También podemos realizar una visualización similar pero usando Box Plots.
amp_boxplot(av2,
group_by = "species",
tax_show = 20,
tax_aggregate = "Genus",
tax_add = "Phylum",
adjust_zero = T,
plot_log = T) +
scale_color_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f")) +
scale_fill_manual(values = c("#a6cee3", "#b2df8a", "#fdbf6f"))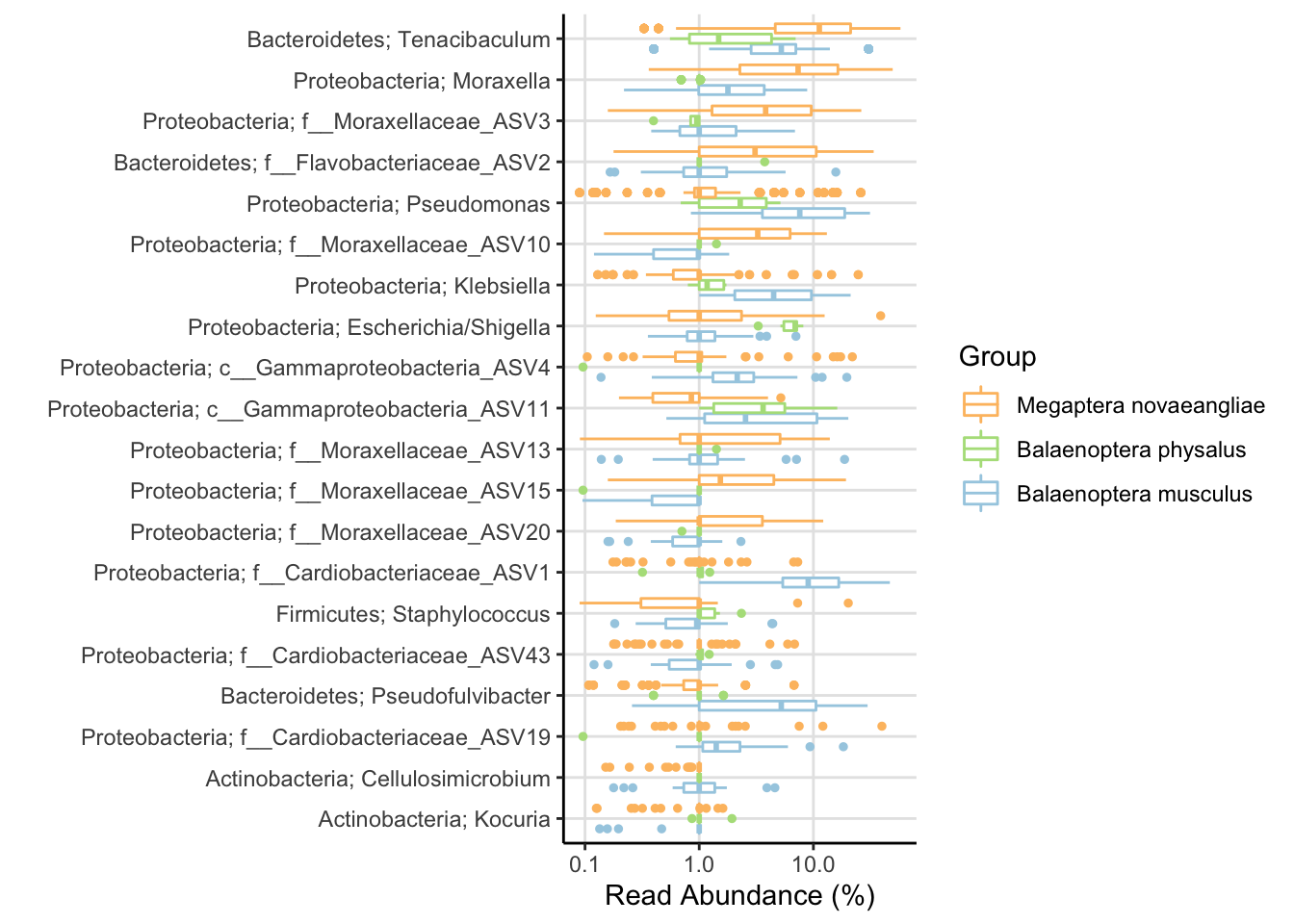
- Veamos ahora si es que algunos de estos microorganismos están compartidos entre todas las muestras. Para esto debemos calcular el core microbiome o el conjunto de taxa compartidas entre un cierto umbral porcentual de muestras y de prevalencia intra-muestra.
- Y visto de otra manera en un diagrama de Venn.
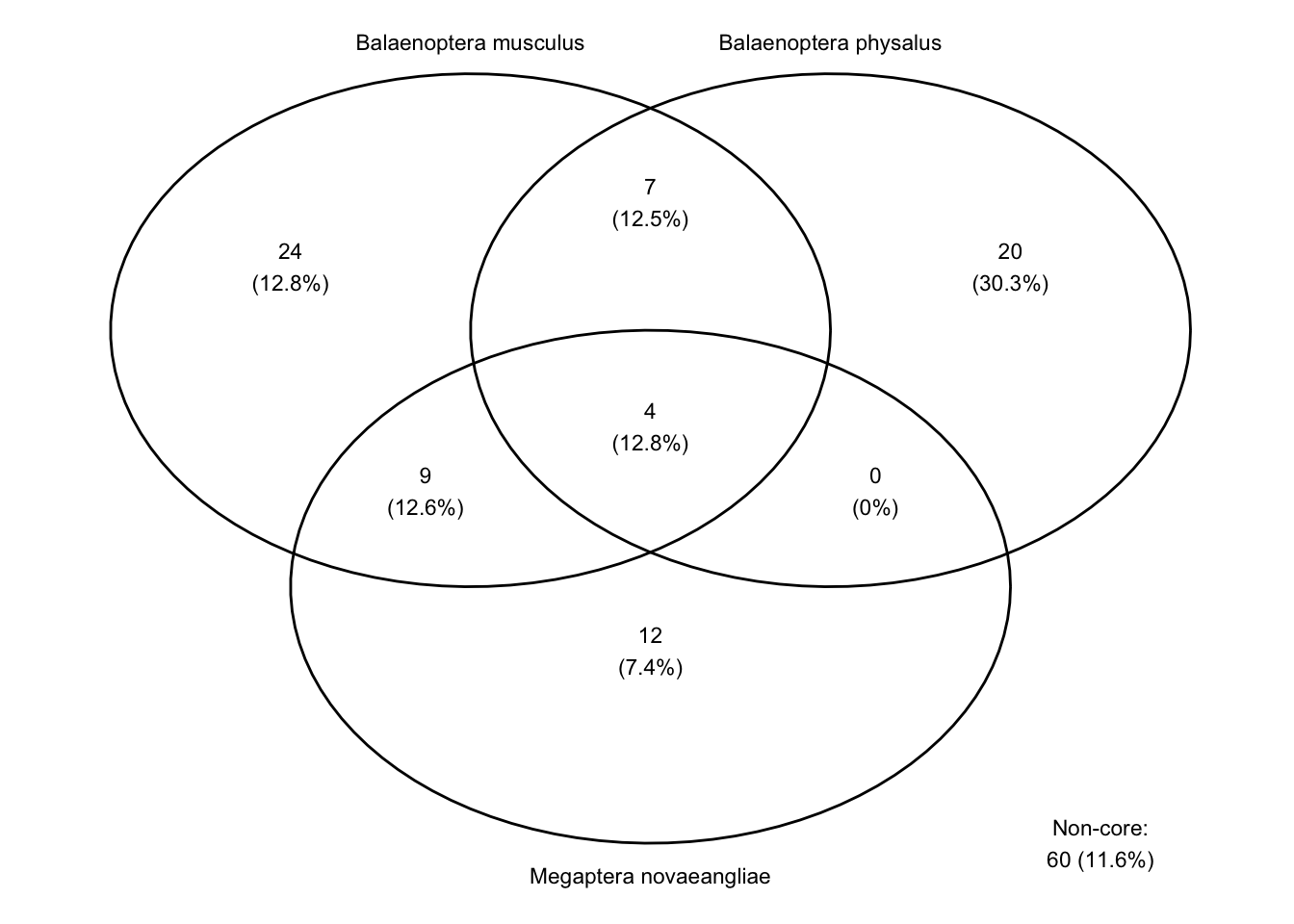
13.3.3 Análisis de abundancia diferencial de microorganismos
Hasta ahora hemos visto principalmente análisis exploratorios y algunos test estadísticos para diversidad alfa y beta. Sin embargo, muchas veces queremos determinar exactamente qué taxa está más representada en una condición versus otra y en qué medida. El procedimiento es análogo al análisis de expresión diferencial de genes en transcritómica, e.g., RNA-seq. Es tan así que justamente ocupamos uno de los paquetes de R más populares en transcriptómica, DESeq2. Ahora, nuestras muestras se secuenciaron al mismo tiempo y se intentó que se produjera una profundidad uniforme a través de todas las muestras. En la práctica esto no ocurre y al momento de analizar las muestras en el contexto del análisis diferencial de abundancia debemos corregir por dos situaciones: el tamaño desigual de las muestras (en número de reads) y la correlación de la variabilidad de los datos con la media de éstos. Para esto último, utilizamos una transformación llamada Variance Stabilizing Transformation, cuyo objetivo es encontrar una función ƒ que se aplique a los valores de read counts, de tal forma que en los nuevos valores y = ƒ(x), la variabilidad de y no se relacione con sus valores medios.
- Comencemos por aplicar la transformación en los datos.
# Creamos un objeto DESeq2 con la función `phyloseq_to_deseq2`
diagdds = phyloseq_to_deseq2(psd4, ~species)
# Calculamos los factores de tamaño como parte de la normalización de las muestras
# calculate geometric means prior to estimate size factors
gm_mean = function(x, na.rm=TRUE){
exp(sum(log(x[x > 0]), na.rm=na.rm) / length(x))
}
geoMeans = apply(counts(diagdds), 1, gm_mean)
diagdds = estimateSizeFactors(diagdds, geoMeans = geoMeans)
# Normalizamos y realizamos el test paramétrico de Wald para determinar taxa diferencialmente abundante.
diagdds = DESeq(diagdds, test="Wald", fitType="local")Hasta ahora hemos transformado nuestro objeto phyloseq en un objeto DESeq2 de nombre diagdds, y hemos normalizado las cuentas y realizado un test paramétrico (Wald Test).
- Nos queda entonces revisar los resultados usando la función
results.
# Guardamos los resultados en el objeto res
res = results(diagdds, cooksCutoff = FALSE)
# hacemos un poco de aseo y ordenamos la tabla de resultados según p-value, y dejamos los valores NA al final
res = res[order(res$padj, na.last=NA), ]- Ahora nosotros queremos averiguar sobre ciertos contrastes específicos entre condiciones, e.g., ballena jorobada versus ballena azul. En el contraste, pasamos un vector con el nombre de la columna en la metadata (“species”) e indicamos el numerador de la comparación (“Megaptera novaeangliae”) y el denominador (“Balaenoptera musculus”). Por lo tanto el
log2FoldChangepositivo indicará que ese microorganismo es más abundante en ballena jorobada que en ballena azul, y viceversa.
res.joro.azul <- results(diagdds, contrast=c("species","Megaptera novaeangliae","Balaenoptera musculus"))# Veamos qué hay en res.joro.azul
res.joro.azul %>%
kable(format = "html", col.names = colnames(res.joro.azul)) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "400px") | baseMean | log2FoldChange | lfcSE | stat | pvalue | padj | |
|---|---|---|---|---|---|---|
| ASV1 | 2719.4817399 | -7.1224534 | 1.0359380 | -6.8753665 | 0.0000000 | 0.0000000 |
| ASV2 | 2022.5405509 | 5.4006313 | 0.7461453 | 7.2380427 | 0.0000000 | 0.0000000 |
| ASV3 | 2100.9914000 | 5.3802668 | 0.6766070 | 7.9518346 | 0.0000000 | 0.0000000 |
| ASV4 | 489.6362991 | 0.8724453 | 0.8271381 | 1.0547760 | 0.2915278 | 0.3442805 |
| ASV5 | 1138.1509390 | 13.0147565 | 1.3019415 | 9.9964222 | 0.0000000 | 0.0000000 |
| ASV6 | 45.6810800 | 5.5840818 | 1.8653474 | 2.9935880 | 0.0027572 | 0.0049735 |
| ASV7 | 245.1490808 | 4.4924803 | 0.9791466 | 4.5881589 | 0.0000045 | 0.0000168 |
| ASV8 | 1211.9442253 | -3.2250402 | 0.9315225 | -3.4621172 | 0.0005359 | 0.0012307 |
| ASV9 | 148.8626474 | 4.0117257 | 0.8333867 | 4.8137625 | NA | NA |
| ASV10 | 548.7130326 | 6.4054200 | 0.7366579 | 8.6952434 | 0.0000000 | 0.0000000 |
| ASV11 | 1009.5925018 | -5.0854806 | 0.8255799 | -6.1598889 | 0.0000000 | 0.0000000 |
| ASV12 | 658.7876904 | -3.2447714 | 1.0411077 | -3.1166530 | 0.0018292 | 0.0034895 |
| ASV13 | 410.6582559 | 3.0099935 | 0.8716746 | 3.4531160 | 0.0005542 | 0.0012494 |
| ASV14 | 140.0717828 | 3.8198888 | 1.0080979 | 3.7892044 | NA | NA |
| ASV15 | 379.3194323 | 7.5644546 | 0.8161418 | 9.2685548 | 0.0000000 | 0.0000000 |
| ASV16 | 285.7620345 | 7.6440491 | 0.9097626 | 8.4022455 | 0.0000000 | 0.0000000 |
| ASV17 | 379.4487960 | 14.2183560 | 1.4003173 | 10.1536672 | 0.0000000 | 0.0000000 |
| ASV18 | 203.0595008 | 9.1750665 | 1.1303913 | 8.1167175 | 0.0000000 | 0.0000000 |
| ASV19 | 69.8158128 | -2.2614994 | 0.9908843 | -2.2823041 | 0.0224714 | 0.0331721 |
| ASV20 | 299.9612618 | 5.6657701 | 0.9446883 | 5.9975021 | 0.0000000 | 0.0000000 |
| ASV21 | 91.1653741 | 7.8577937 | 1.1458691 | 6.8574966 | 0.0000000 | 0.0000000 |
| ASV22 | 522.4807634 | -3.8585579 | 1.1993634 | -3.2171718 | 0.0012946 | 0.0027209 |
| ASV23 | 100.3448777 | 10.0985821 | 1.3550063 | 7.4527934 | 0.0000000 | 0.0000000 |
| ASV24 | 345.5275036 | 8.1857232 | 1.3845014 | 5.9123979 | 0.0000000 | 0.0000000 |
| ASV25 | 278.9908685 | -2.5125876 | 1.5875681 | -1.5826644 | 0.1134980 | 0.1436097 |
| ASV26 | 62.1557348 | -0.4907744 | 1.4756983 | -0.3325709 | 0.7394582 | 0.7904553 |
| ASV27 | 189.6100970 | -3.6035868 | 1.2050796 | -2.9903310 | 0.0027868 | 0.0049735 |
| ASV28 | 218.1259662 | -0.4960691 | 0.8838224 | -0.5612769 | 0.5746088 | 0.6361740 |
| ASV29 | 348.5666726 | -9.2693864 | 1.2067447 | -7.6813154 | 0.0000000 | 0.0000000 |
| ASV30 | 19.9949713 | -6.9404408 | 2.0012541 | -3.4680458 | NA | NA |
| ASV31 | 9.4574754 | 4.4204992 | 1.8265012 | 2.4202006 | 0.0155119 | 0.0234571 |
| ASV32 | 135.9042193 | 4.6452237 | 1.0957355 | 4.2393659 | 0.0000224 | 0.0000678 |
| ASV33 | 12.2150782 | 3.8067681 | 1.2271358 | 3.1021570 | NA | NA |
| ASV34 | 14.5758192 | 6.9563749 | 0.9863495 | 7.0526474 | 0.0000000 | 0.0000000 |
| ASV35 | 78.7642402 | 1.0289747 | 0.8831016 | 1.1651827 | NA | NA |
| ASV36 | 15.6844519 | 4.4745682 | 1.5457245 | 2.8948031 | 0.0037940 | 0.0065340 |
| ASV37 | 104.0931206 | 6.3198854 | 1.0890983 | 5.8028606 | 0.0000000 | 0.0000000 |
| ASV38 | 63.0999441 | 7.0198666 | 1.2124308 | 5.7899111 | 0.0000000 | 0.0000000 |
| ASV39 | 71.6857366 | -4.1210982 | 0.9699437 | -4.2488015 | 0.0000215 | 0.0000666 |
| ASV40 | 11.7563573 | 4.6362327 | 1.8491255 | 2.5072570 | NA | NA |
| ASV41 | 63.6924912 | 6.7765578 | 1.5717381 | 4.3115057 | 0.0000162 | 0.0000522 |
| ASV42 | 39.8228286 | 3.9641573 | 1.1704109 | 3.3869791 | 0.0007067 | 0.0015108 |
| ASV43 | 29.6927766 | 0.6482185 | 0.9863497 | 0.6571893 | 0.5110592 | 0.5761031 |
| ASV44 | 4.6851145 | -4.9032274 | 1.1881480 | -4.1267818 | 0.0000368 | 0.0001061 |
| ASV45 | 31.3542359 | -5.8903163 | 1.0674036 | -5.5183589 | 0.0000000 | 0.0000001 |
| ASV46 | 52.3468950 | 8.1940044 | 1.2045392 | 6.8026052 | 0.0000000 | 0.0000000 |
| ASV47 | 24.3460080 | 5.1994788 | 1.1498081 | 4.5220406 | 0.0000061 | 0.0000223 |
| ASV48 | 9.5531988 | -3.1476568 | 1.9364467 | -1.6254807 | NA | NA |
| ASV49 | 28.1689872 | 4.3313913 | 1.3563693 | 3.1933718 | 0.0014062 | 0.0028817 |
| ASV50 | 89.6234760 | -8.8288920 | 1.2859692 | -6.8655549 | 0.0000000 | 0.0000000 |
| ASV51 | 42.8236021 | 7.0692754 | 1.5947984 | 4.4327079 | 0.0000093 | 0.0000328 |
| ASV52 | 1.0011767 | 2.5999491 | 1.5148878 | 1.7162651 | 0.0861135 | 0.1112300 |
| ASV53 | 33.2080947 | 1.4605265 | 0.9766925 | 1.4953800 | 0.1348153 | 0.1655158 |
| ASV54 | 82.8130820 | -2.1815886 | 1.1338739 | -1.9240134 | 0.0543529 | 0.0732583 |
| ASV55 | 9.1745027 | 3.2672435 | 1.9477963 | 1.6774051 | 0.0934633 | 0.1194789 |
| ASV56 | 11.3403634 | 4.6971790 | 1.4994164 | 3.1326715 | 0.0017322 | 0.0034095 |
| ASV57 | 16.5241450 | 1.8610673 | 1.5605106 | 1.1926015 | 0.2330255 | 0.2778381 |
| ASV58 | 13.2662521 | 2.8954770 | 1.4432286 | 2.0062497 | 0.0448296 | 0.0624592 |
| ASV59 | 99.5242996 | -10.6308795 | 1.2924338 | -8.2254731 | 0.0000000 | 0.0000000 |
| ASV60 | 68.5859180 | 1.1857152 | 1.2128028 | 0.9776653 | 0.3282399 | 0.3839788 |
| ASV61 | 2.5896101 | 0.5237964 | 0.9994077 | 0.5241068 | NA | NA |
| ASV62 | 7.2983417 | 4.2913582 | 1.4469920 | 2.9657096 | 0.0030199 | 0.0052741 |
| ASV63 | 10.6361249 | 7.2294633 | 1.8091772 | 3.9959952 | 0.0000644 | 0.0001816 |
| ASV64 | 83.5838226 | -2.6340549 | 1.2610396 | -2.0887963 | 0.0367261 | 0.0517504 |
| ASV65 | 47.6516393 | 10.1206302 | 1.4127059 | 7.1640036 | 0.0000000 | 0.0000000 |
| ASV66 | 70.2942412 | 5.2089523 | 1.4217267 | 3.6638212 | 0.0002485 | 0.0006032 |
| ASV67 | 24.7841554 | 5.1537365 | 1.2188111 | 4.2284950 | 0.0000235 | 0.0000695 |
| ASV68 | 14.2934620 | 5.5087333 | 1.4218510 | 3.8743394 | 0.0001069 | 0.0002821 |
| ASV69 | 21.2132029 | 1.7036268 | 1.1444604 | 1.4885853 | 0.1365966 | 0.1660586 |
| ASV70 | 68.7558900 | -0.3460662 | 1.6848409 | -0.2053999 | 0.8372597 | 0.8516169 |
| ASV71 | 6.2303015 | -0.2601080 | 1.2445510 | -0.2089974 | NA | NA |
| ASV72 | 14.6638119 | 7.0648004 | 1.6396906 | 4.3086180 | 0.0000164 | 0.0000522 |
| ASV73 | 7.5897409 | 5.3428616 | 1.4510121 | 3.6821619 | 0.0002313 | 0.0005735 |
| ASV74 | 5.0789582 | -1.3853182 | 1.1394033 | -1.2158278 | NA | NA |
| ASV75 | 19.8697343 | 24.2907265 | 1.8316754 | 13.2614802 | 0.0000000 | 0.0000000 |
| ASV76 | 1.2239068 | -1.9354764 | 1.2311449 | -1.5720947 | 0.1159286 | 0.1437514 |
| ASV77 | 9.0374513 | -9.8994430 | 1.4981683 | -6.6076975 | 0.0000000 | 0.0000000 |
| ASV78 | 21.1929292 | 8.8483467 | 1.6584029 | 5.3354626 | 0.0000001 | 0.0000004 |
| ASV79 | 13.7751655 | -2.6546117 | 1.4029815 | -1.8921217 | 0.0584748 | 0.0771369 |
| ASV80 | 28.3678395 | 23.0655138 | 1.6147380 | 14.2843691 | 0.0000000 | 0.0000000 |
| ASV81 | 9.9644103 | -2.8946247 | 0.7983325 | -3.6258383 | NA | NA |
| ASV82 | 5.5165903 | 5.0158379 | 1.8121294 | 2.7679247 | 0.0056414 | 0.0095827 |
| ASV83 | 22.0290096 | 7.8115706 | 1.2982118 | 6.0171773 | 0.0000000 | 0.0000000 |
| ASV84 | 5.3880867 | 3.5536543 | 1.1892872 | 2.9880540 | 0.0028076 | 0.0049735 |
| ASV85 | 1.7369590 | -3.9773770 | 1.3253471 | -3.0010078 | 0.0026909 | 0.0049735 |
| ASV86 | 8.6940194 | 0.9909398 | 1.7309680 | 0.5724772 | 0.5669987 | 0.6334040 |
| ASV87 | 2.6463367 | -4.9527209 | 1.2607597 | -3.9283623 | 0.0000855 | 0.0002305 |
| ASV88 | 22.4846884 | -7.9573124 | 1.0142627 | -7.8454155 | 0.0000000 | 0.0000000 |
| ASV89 | 29.2974383 | -5.7553117 | 1.0928471 | -5.2663465 | 0.0000001 | 0.0000005 |
| ASV90 | 5.5849388 | 4.5854927 | 2.0669960 | 2.2184332 | 0.0265253 | 0.0386957 |
| ASV91 | 16.5698452 | -7.4960519 | 1.3749392 | -5.4519154 | 0.0000000 | 0.0000002 |
| ASV92 | 22.6522435 | -4.9676859 | 1.2977662 | -3.8278742 | 0.0001293 | 0.0003339 |
| ASV93 | 6.4062042 | -4.0851822 | 1.5797024 | -2.5860455 | 0.0097084 | 0.0156343 |
| ASV94 | 1.7970262 | 3.0983231 | 1.2328023 | 2.5132359 | 0.0119629 | 0.0190180 |
| ASV95 | 11.9501380 | -7.6996523 | 1.3118837 | -5.8691576 | 0.0000000 | 0.0000000 |
| ASV96 | 2.8351525 | 0.3828029 | 1.6800387 | 0.2278536 | 0.8197600 | 0.8470854 |
| ASV97 | 3.7248506 | 5.1135350 | 1.5097237 | 3.3870667 | 0.0007064 | 0.0015108 |
| ASV98 | 4.3233609 | 4.8042120 | 1.9622148 | 2.4483619 | 0.0143507 | 0.0219690 |
| ASV99 | 2.8243846 | 3.5024621 | 1.0975926 | 3.1910403 | 0.0014176 | 0.0028817 |
| ASV100 | 9.9778511 | -4.6282742 | 1.2648220 | -3.6592297 | 0.0002530 | 0.0006032 |
| ASV101 | 7.1881927 | -0.4163206 | 1.6084949 | -0.2588262 | 0.7957694 | 0.8292050 |
| ASV102 | 8.2413114 | -3.7473291 | 1.1989296 | -3.1255621 | 0.0017747 | 0.0034384 |
| ASV103 | 8.3102765 | -6.7562778 | 1.5258751 | -4.4278053 | 0.0000095 | 0.0000328 |
| ASV104 | 6.7857308 | 5.7064612 | 1.6741681 | 3.4085353 | 0.0006531 | 0.0014462 |
| ASV105 | 5.0488641 | 4.1346140 | 1.5935323 | 2.5946219 | 0.0094695 | 0.0154502 |
| ASV106 | 0.9541234 | -1.1028164 | 2.0346151 | -0.5420270 | 0.5877999 | 0.6450193 |
| ASV107 | 4.1007567 | 4.9980913 | 1.1373183 | 4.3946286 | 0.0000111 | 0.0000372 |
| ASV108 | 5.4038096 | -3.6271094 | 1.2133551 | -2.9893224 | 0.0027960 | 0.0049735 |
| ASV109 | 1.3501927 | -0.4206071 | 1.4415801 | -0.2917681 | 0.7704639 | 0.8096400 |
| ASV110 | 2.7371281 | 3.3005287 | 1.7374702 | 1.8996174 | 0.0574833 | 0.0766445 |
| ASV111 | 2.4259080 | 4.5000589 | 1.8203672 | 2.4720611 | 0.0134337 | 0.0210857 |
| ASV112 | 1.5325388 | -0.6703858 | 1.9366425 | -0.3461588 | 0.7292234 | 0.7862930 |
| ASV113 | 5.1286581 | 5.5651234 | 1.5523269 | 3.5850202 | 0.0003371 | 0.0007886 |
| ASV114 | 3.2084055 | 4.2076848 | 1.5538169 | 2.7079669 | 0.0067697 | 0.0113438 |
| ASV115 | 1.8693006 | 4.1459636 | 1.5967342 | 2.5965270 | 0.0094171 | 0.0154502 |
| ASV116 | 7.7258899 | -6.1720272 | 1.5544558 | -3.9705388 | 0.0000717 | 0.0001976 |
| ASV117 | 4.6721853 | -6.3099610 | 1.6600099 | -3.8011586 | 0.0001440 | 0.0003645 |
| ASV118 | 0.9186556 | -1.6434260 | 1.7179194 | -0.9566375 | 0.3387503 | 0.3925704 |
| ASV119 | 7.0228498 | -1.8945403 | 0.8629677 | -2.1953780 | 0.0281365 | 0.0401026 |
| ASV120 | 0.8432035 | 0.1593532 | 1.9188207 | 0.0830475 | 0.9338138 | 0.9338138 |
| ASV121 | 3.3482616 | -2.7839431 | 1.2669338 | -2.1973864 | 0.0279929 | 0.0401026 |
| ASV122 | 3.6359471 | -4.5928491 | 1.8643476 | -2.4635155 | 0.0137582 | 0.0213252 |
| ASV123 | 1.7902754 | -1.4023022 | 1.5327540 | -0.9148906 | 0.3602491 | 0.4136193 |
| ASV124 | 2.6697932 | 2.1839877 | 1.3857261 | 1.5760602 | 0.1150119 | 0.1437514 |
| ASV125 | 1.1415391 | -0.7290996 | 1.8509376 | -0.3939082 | 0.6936488 | 0.7544952 |
| ASV126 | 2.9465902 | -1.8431157 | 1.2640321 | -1.4581241 | 0.1448064 | 0.1743300 |
| ASV127 | 2.9109028 | 0.9547596 | 1.1013290 | 0.8669159 | 0.3859881 | 0.4391057 |
| ASV128 | 2.9624475 | -4.1618781 | 1.3116973 | -3.1728951 | 0.0015093 | 0.0030185 |
| ASV129 | 2.1092384 | -0.3088813 | 1.5096505 | -0.2046045 | 0.8378811 | 0.8516169 |
| ASV130 | 1.4749075 | -0.2884521 | 0.9376518 | -0.3076324 | 0.7583621 | 0.8037342 |
| ASV131 | 2.3920165 | -2.4765750 | 1.3681047 | -1.8102232 | 0.0702612 | 0.0917093 |
| ASV132 | 1.3600064 | 0.1650124 | 1.5324853 | 0.1076763 | 0.9142524 | 0.9216854 |
| ASV133 | 0.7154383 | 2.8063290 | 1.4139574 | 1.9847338 | 0.0471741 | 0.0649954 |
| ASV134 | 1.0638791 | -1.5182223 | 1.3665996 | -1.1109489 | NA | NA |
| ASV135 | 1.4368076 | -4.0800926 | 1.6943303 | -2.4080857 | 0.0160364 | 0.0239580 |
| ASV136 | 0.6620716 | -2.3480281 | 1.2061752 | -1.9466725 | 0.0515740 | 0.0702767 |
¿Qué significa cada columna? Revisa la viñeta de DESeq2 aquí.
- Ahora establezcamos un umbral de significancia estadística para los valores de p-value ajustado o
padj. Cualquier resultado bajo este umbral será considerado no significativo y viceversa.
# Este es nuestro umbral
alpha = 0.01
# Ordenamos la tabla de resultados
res.joro.azul = res.joro.azul[order(res.joro.azul$padj, na.last=NA), ]
# Filtramos según nuestro umbral alpha
sigtab = res.joro.azul[(res.joro.azul$padj < alpha), ]
# Le agregamos la taxonomía a la tabla
sigtab = cbind(as(sigtab, "data.frame"), as(tax_table(psd5)[rownames(sigtab), ], "matrix"))
# Manipulaciones varias para finalmente graficar los resultados
sigtabgen = subset(sigtab, !is.na(Genus))
# Phylum order
x = tapply(sigtabgen$log2FoldChange, sigtabgen$Phylum, function(x) max(x))
x = sort(x, TRUE)
sigtabgen$Phylum = factor(as.character(sigtabgen$Phylum), levels=names(x))
# Genus order
x = tapply(sigtabgen$log2FoldChange, sigtabgen$Genus, function(x) max(x))
x = sort(x, TRUE)
sigtabgen$Genus = factor(as.character(sigtabgen$Genus), levels=names(x))
ggplot(sigtabgen, aes(y=Genus, x=log2FoldChange, color=Phylum)) +
geom_vline(xintercept = 0.0, color = "gray", size = 0.5) +
geom_point(size=4) +
theme(axis.text.x = element_text(angle = -90, hjust = 0, vjust=0.5, size = 10), axis.text.y = element_text(size = 13), legend.text = element_text(size = 13) )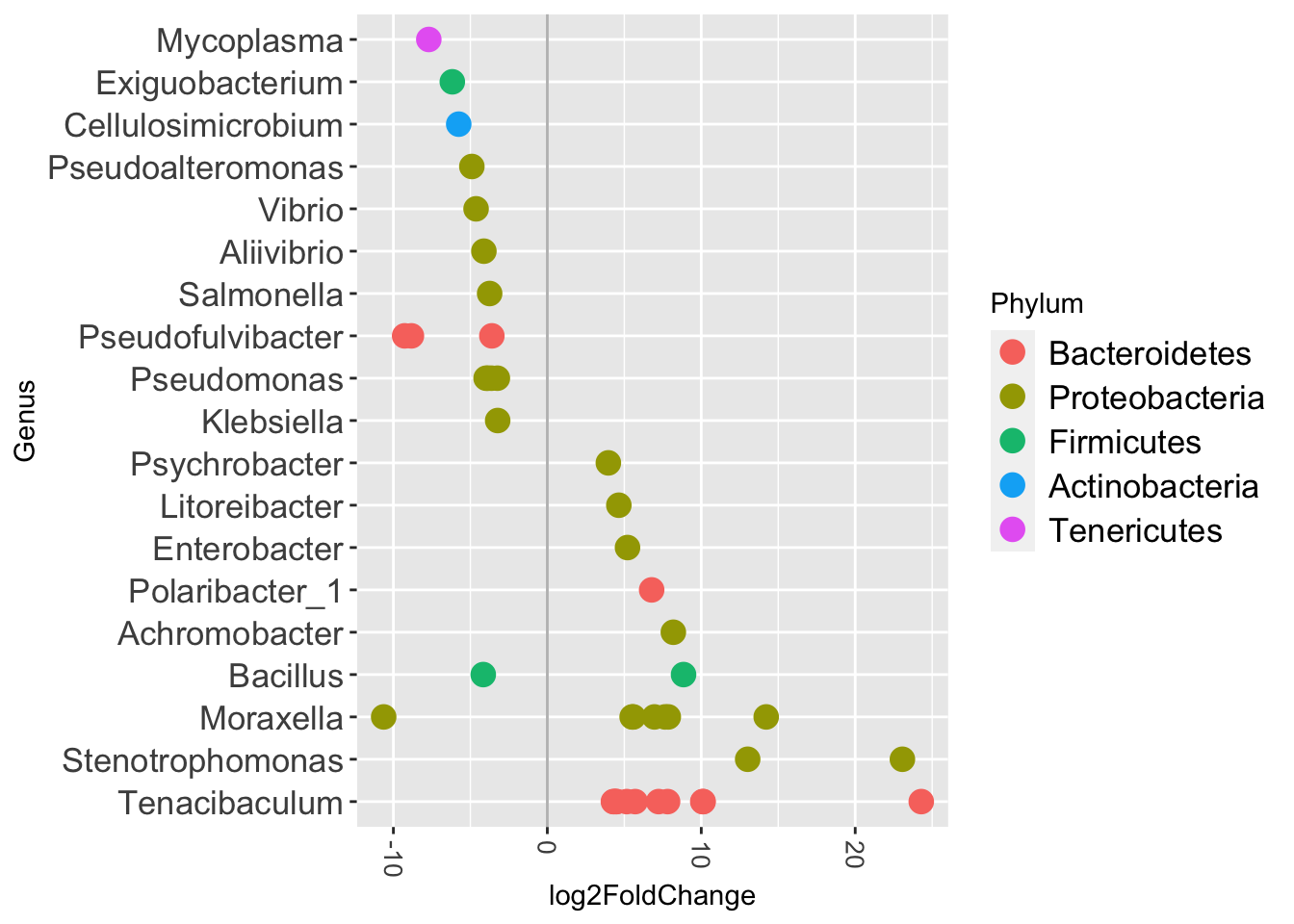
13.3.4 Redes de co-ocurrencia
Para finalizar, vamos a echar un vistazo a las capacidades de phyloseq para generar redes de co-occurrencia. Las redes de co-ocurrencia nos dan pistas sobre potenciales interacciones ecológicas entre organismos. Estas interacciones pueden ser directas o indirectas (no lo podemos determinar a partir de una red) y nos permiten comenzar a descifrar mecanismos ecológicos detrás de la composición de una comunidad microbiana. En general en ecología tenemos distintos tipos de interacciones:

Donde destacan depredación, competición o depredación mutua, y mutualismo. Cada una de estas relaciones podría ser detectada en una red de co-ocurrencia según patrones de correlación positivos o negativos.
- Veamos como generaríamos una red en
phyloseq.
plot_net(psd5, type = "taxa", point_label = "Genus", point_size = 10, point_alpha = 0.5, maxdist = 0.5, color = "Phylum", distance = "bray", laymeth = "auto") 
La red generada con phyloseq no es una red de co-ocurrencia propiamente tal. Es más bien una red que representa relaciones de distancia entre taxa o muestras. En nuestro ejemplo usamos muestras. Para una red de co-ocurrencia propiamente tal necesitamos usar las funciones del paquete SpiecEasi.
- Veamos un ejemplo de cómo hacerlo.
library(devtools)
# install_github("zdk123/SpiecEasi")
library(SpiecEasi)
se.mb.psd4 <- spiec.easi(psd4, method='mb', lambda.min.ratio=1e-2,
nlambda=20, icov.select.params=list(rep.num=50))
ig2.mb <- adj2igraph(getRefit(se.mb.psd4), vertex.attr=list(name=taxa_names(psd4)))
plot_network(ig2.mb, psd4, type='taxa', color="Phylum")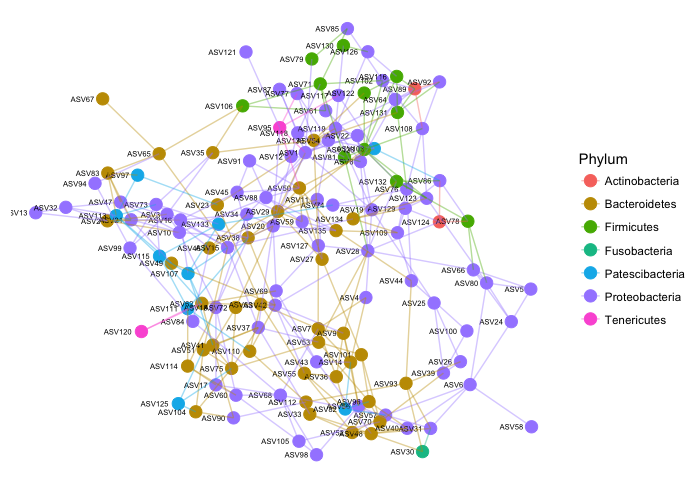
Un tutorial mucho más completo lo pueden encontrar aquí y aquí.