12 Introducción a phyloseq
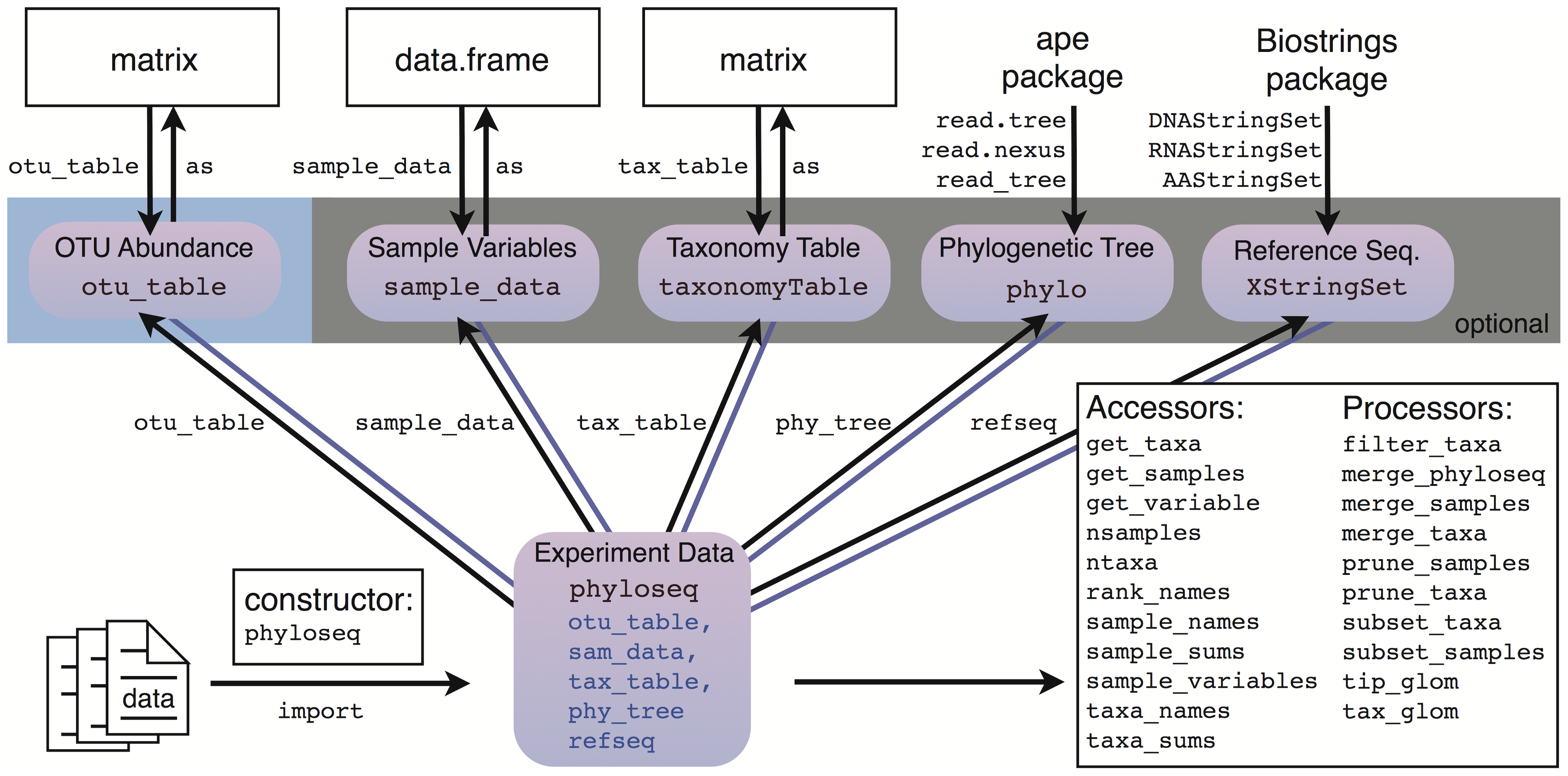
Phyloseq es un paquete de Bioconductor (Open Source Software For Bioinformatics) para la manipulación y análisis de datos metagenómicos generados por metodologías de secuenciación de alto rendimiento. phyloseq es una herramienta para importar, guardar, analizar y visualizar éste tipo de datos después de haber sido procesados inicialmente, e.g., ensamblaje de novo, ASVs u OTUs (clustered), incluyendo otros importantes datos asociados (si están disponibles): tabla de observaciones asociadas a cada muestra (e.g., especie, localización geográfica, temperatura, etc.), conocida como sample data o metadata, árbol filogenético, e identificación taxonómica de cada OTU. La estructura del paquete phyloseq consiste en una serie de funciones de acceso y de proceso de objetos phyloseq. Estos objetos están compuestos de cuatro componentes que almacenan las cuentas de reads, la metadata, la taxonomía y el árbol filogenético. El paquete también provee una serie de herramientas para importar datos de otros programas. El siguiente diagrama muestra la estructura completa de phyloseq.
Hasta este punto tenemos dos análisis, uno con la pipeline DADA2 (basado en variantes de secuencia) y otro con mothur (basado en OTUs).
- En la terminal de R carguemos ambos resultados:
# Leémos el objeto phyloseq del análisis por DADA2
library(phyloseq)
(readRDS("data/ps.RDS") -> psd)## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 1476 taxa and 98 samples ]
## sample_data() Sample Data: [ 98 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 1476 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 1476 tips and 1474 internal nodes ]## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 2696 taxa and 98 samples ]
## sample_data() Sample Data: [ 98 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 2696 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 2696 tips and 2695 internal nodes ]Lo primero que salta a la vista es que ambos análisis identificaron un número diferente de unidades taxonómicas, i.e., 1476 vs. 2696. El resultado es un tanto esperado considerado que es sabido que al formar OTUs se tiende a sobre estimar la riqueza, i.e., el número de unidades taxonómicas, en una muestra.
Ahora, en ambos casos probablemente tenemos muchas OTUs que corresponden a falsos positivos las cuales pueden ser identificadas y filtradas antes de hacer cualquier análisis serio.
12.1 Control de calidad del análisis de 16S
Lo primero que podemos mirar es la prevalencia de las features taxonómicas.
- Primero creamos un data frame con los valores de prevalencia, luego les agregamos la taxonomía y graficamos.
# Computamos prevalencia para cada feature y la guardamos en un data frame
prevdf = apply(X = otu_table(psd),
MARGIN = ifelse(taxa_are_rows(psd), yes = 1, no = 2),
FUN = function(x){sum(x > 0)})
# Le agregamos la taxonomía
prevdf = data.frame(Prevalence = prevdf,
TotalAbundance = taxa_sums(psd),
tax_table(psd))
plyr::ddply(prevdf, "Phylum", function(df1){cbind(mean(df1$Prevalence),sum(df1$Prevalence))}) -> dfprev
kable(dfprev)| Phylum | 1 | 2 |
|---|---|---|
| Acidobacteria | 3.800000 | 38 |
| Actinobacteria | 5.041667 | 363 |
| Bacteroidetes | 5.719388 | 2242 |
| BRC1 | 1.000000 | 1 |
| Chloroflexi | 1.750000 | 21 |
| Ciliophora | 3.000000 | 9 |
| Cyanobacteria | 4.223881 | 283 |
| Deinococcus-Thermus | 1.333333 | 4 |
| Epsilonbacteraeota | 2.760000 | 69 |
| Euryarchaeota | 3.714286 | 26 |
| Firmicutes | 4.685315 | 670 |
| Fusobacteria | 3.500000 | 70 |
| Gemmatimonadetes | 1.000000 | 2 |
| Kiritimatiellaeota | 1.000000 | 1 |
| Lentisphaerae | 1.250000 | 5 |
| Marinimicrobia_(SAR406_clade) | 3.500000 | 7 |
| Nanoarchaeaeota | 1.000000 | 1 |
| Nitrospinae | 1.000000 | 1 |
| Ochrophyta | 1.000000 | 1 |
| Patescibacteria | 5.473684 | 312 |
| Planctomycetes | 3.235294 | 110 |
| Proteobacteria | 8.233840 | 4331 |
| Schekmanbacteria | 1.000000 | 1 |
| Spirochaetes | 1.500000 | 3 |
| Tenericutes | 6.083333 | 73 |
| Thaumarchaeota | 4.000000 | 12 |
| Verrucomicrobia | 4.814815 | 130 |
| NA | 6.425532 | 302 |
Al examinar la tabla, es evidente que algunos Phylum aunque presentes, están muy poco representados. La columna 1 representa la media de read counts para ese Phylum, mientras que la columna 2 representa la suma. Por ejemplo, grupos como BRC1, Kiritimatiellaeota, y Nanoarchaeaeota están representados solamente por 1 read. Es muy riesgoso mantener estos grupos taxonómicos en el análisis ya que pueden corresponder a falsos positivos.
- Para filtrarlos, generamos un vector con todas las taxa que queremos filtrar.
# Definimos taxa a filtrar
filterPhyla = c("BRC1", "Deinococcus-Thermus", "Gemmatimonadetes", "Kiritimatiellaeota", "Nanoarchaeaeota", "Ochrophyta", "Schekmanbacteria", "Ciliophora", "Spirochaetes", NA)
# Procedemos a filtrar
(psd1 = subset_taxa(psd, !Phylum %in% filterPhyla))## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 1414 taxa and 98 samples ]
## sample_data() Sample Data: [ 98 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 1414 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 1414 tips and 1412 internal nodes ]# Además aprovechamos a remover taxa que no corresponde a microorganismos como cloroplastos, mitocondrias y otros
filterPhyla2 <- c("Chloroplast", "Mitochondria", "Eukaryota")
psd1 <- subset_taxa(psd1, !Kingdom %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Phylum %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Class %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Order %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Family %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Genus %in% filterPhyla2)- Además del filtrado que acabamos de realizar, existen otros tipos de filtrado que tienen que ver con la media de read counts por taxa, con la distribución de éstas, y con filtrar muestras bajo un número mínimo de reads.
# Filtramos taxa de acuerdo a un umbral de número medio de _read counts_, en este caso 1e-5
psd2 <- filter_taxa(psd1, function(x) mean(x) > 1e-5, TRUE)
# También podemos remover taxa que no se observe más de X veces en al menos 10% de las muestras
psd3 <- filter_taxa(psd2, function(x) sum(x > 2) > (0.1*length(x)), TRUE)
# Y finalmente filtrar muestras con menos de 1000 reads
psd4 = prune_samples(sample_sums(psd3) > 1000, psd3)
psd4## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]- Otra forma de filtrar taxa de baja prevalencia es estableciendo un umbral y luego visulizar el efecto de manera grafica.
# Seleccionamos las taxa de interés
prevdf1 = subset(prevdf, Phylum %in% get_taxa_unique(psd4, "Phylum"))
ggplot(prevdf1, aes(TotalAbundance, Prevalence / nsamples(psd),color=Phylum)) +
# Agregamos una línea para nuestro umbral
geom_hline(yintercept = 0.05, alpha = 0.5, linetype = 2) + geom_point(size = 2, alpha = 0.7) +
scale_x_log10() + xlab("Total Abundance") + ylab("Prevalence [Frac. Samples]") +
facet_wrap(~Phylum) + theme(legend.position="none")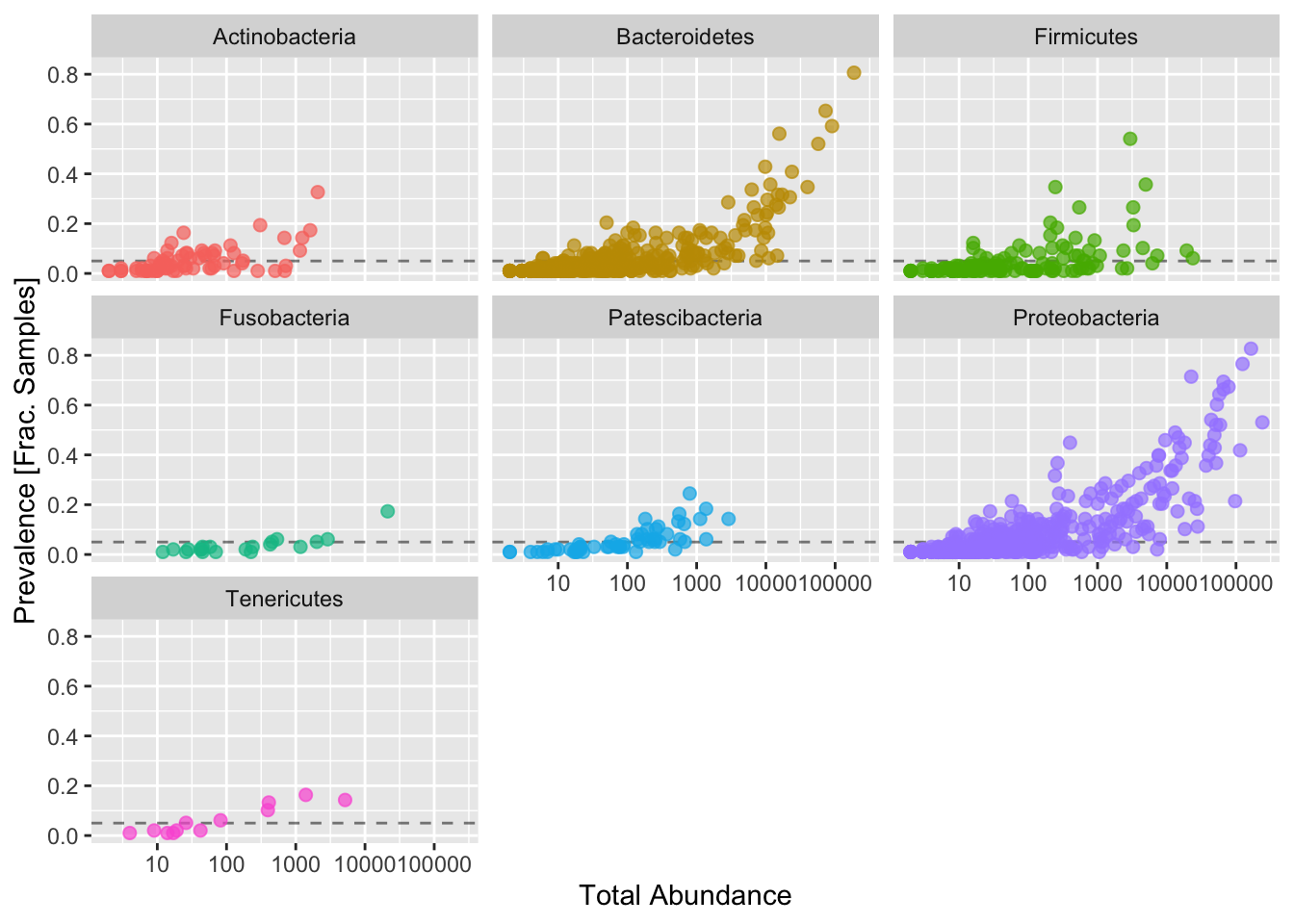
## [1] 4.35# Execute prevalence filter, using `prune_taxa()` function
keepTaxa = rownames(prevdf1)[(prevdf1$Prevalence >= prevalenceThreshold)]
(psd5 = prune_taxa(keepTaxa, psd4))## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]DADA2 usa como nombre de las taxa la secuencia o ASV asociada a un taxon determinado. Esto es conveniente cuando nos interesa la secuencia en nuestros análisis, sin embargo en este práctico solamente vamos a trabajar a nivel de comunidad.
- Por esto, vamos a reemplazar esos nombres con códigos correlativos, lo cual va a hacer las visualizaciones posteriores más entendibles.
# Reemplazamos las secuencias por un nombre genérico
taxa_names(psd5) <- paste0("ASV", seq(ntaxa(psd5)))- Con nuestro objeto phyloseq ya filtrado y listo para usar, podemos gráficar la distribución de read counts por número de muestra de forma de tener una idea sobre la distribución de éstas.
sample_sum_df <- data.frame(sum = sample_sums(psd5))
ggplot(sample_sum_df, aes(x = sum)) +
geom_histogram(color = "black", fill = "grey", binwidth = 2500) +
ggtitle("Distribution of sample sequencing depth") +
xlab("Read counts") +
theme(axis.title.y = element_blank()) 
- Finalmente, calculamos curvas de rarefacción para cada muestra, de manera tal que podamos determinar si la profundidad de secuenciación fue sufuciente o si tal vez necesitemos secuenciar más. En otras palabras, este análisis nos permitiría averiguar si al secuenciar más observaríamos más OTUs o ASVs.
# Primero cargamos algunos scripts de manera remota
scripts <- c("graphical_methods.R",
"tree_methods.R",
"plot_merged_trees.R",
"specificity_methods.R",
"ternary_plot.R",
"richness.R",
"edgePCA.R",
"copy_number_correction.R",
"import_frogs.R",
"prevalence.R",
"compute_niche.R")
urls <- paste0("https://raw.githubusercontent.com/mahendra-mariadassou/phyloseq-extended/master/R/", scripts)
for (url in urls) {
source(url)
}# Y graficamos
p <- ggrare(psd5, step = 100, color = "species", label = "sample_ID", se = TRUE)
(p <- p + facet_wrap(~species))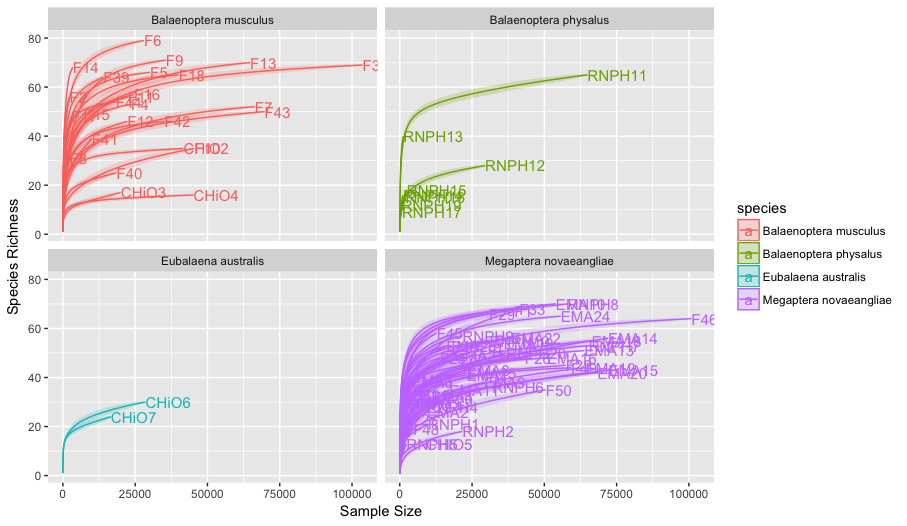
Los gráficos están separados por especies de ballena, azul (Balaenoptera musculus), fin (Balaenoptera physalus), franca (Eubalaena australis), y jorobada (Megaptera novaeangliae) y muestran la cantidad de Taxa (riqueza o diversidad alfa) en función del tamaño muestreal o número de reads. Podemos ver que la mayoría de las muestras de ballena azul y jorobada alcanzan un plateau. Esto significa que el retorno en la inversión disminuye si seguimos secuenciando, o de otra forma, que ya hemos muestreado toda la diversidad de esa muestra. Al contrario, algunas muestras de ballena fin no alcanzaron el plateau, lo cual implica que la diversidad alfa estaría subestimada.
12.2 Estructura y manipulación de un objeto phyloseq
Muchas veces queremos analizar un sub conjunto de las muestras en nuestro objeto phyloseq, o bien, queremos seleccionar ciertos grupos taxonómicos para análisis posteriores. phyloseq nos permite hacer todo tipo de filtros para llevar esto a cabo. Veamos dónde se almacena la información en phyloseq.
- Primero la tabla de cuentas.
# Esta es la tabla de cuentas o read counts
head(otu_table(psd5)) %>%
kable(format = "html", col.names = colnames(otu_table(psd5))) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "350px")| ASV1 | ASV2 | ASV3 | ASV4 | ASV5 | ASV6 | ASV7 | ASV8 | ASV9 | ASV10 | ASV11 | ASV12 | ASV13 | ASV14 | ASV15 | ASV16 | ASV17 | ASV18 | ASV19 | ASV20 | ASV21 | ASV22 | ASV23 | ASV24 | ASV25 | ASV26 | ASV27 | ASV28 | ASV29 | ASV30 | ASV31 | ASV32 | ASV33 | ASV34 | ASV35 | ASV36 | ASV37 | ASV38 | ASV39 | ASV40 | ASV41 | ASV42 | ASV43 | ASV44 | ASV45 | ASV46 | ASV47 | ASV48 | ASV49 | ASV50 | ASV51 | ASV52 | ASV53 | ASV54 | ASV55 | ASV56 | ASV57 | ASV58 | ASV59 | ASV60 | ASV61 | ASV62 | ASV63 | ASV64 | ASV65 | ASV66 | ASV67 | ASV68 | ASV69 | ASV70 | ASV71 | ASV72 | ASV73 | ASV74 | ASV75 | ASV76 | ASV77 | ASV78 | ASV79 | ASV80 | ASV81 | ASV82 | ASV83 | ASV84 | ASV85 | ASV86 | ASV87 | ASV88 | ASV89 | ASV90 | ASV91 | ASV92 | ASV93 | ASV94 | ASV95 | ASV96 | ASV97 | ASV98 | ASV99 | ASV100 | ASV101 | ASV102 | ASV103 | ASV104 | ASV105 | ASV106 | ASV107 | ASV108 | ASV109 | ASV110 | ASV111 | ASV112 | ASV113 | ASV114 | ASV115 | ASV116 | ASV117 | ASV118 | ASV119 | ASV120 | ASV121 | ASV122 | ASV123 | ASV124 | ASV125 | ASV126 | ASV127 | ASV128 | ASV129 | ASV130 | ASV131 | ASV132 | ASV133 | ASV134 | ASV135 | ASV136 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SRR6442697 | 0 | 3 | 1 | 4 | 0 | 0 | 5 | 17 | 3 | 0 | 0 | 0 | 1 | 1 | 917 | 1031 | 0 | 3 | 0 | 76 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 | 0 | 0 | 534 | 0 | 4 | 0 | 2099 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 158 | 0 | 914 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1014 | 0 | 0 | 322 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 535 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 122 | 0 | 0 | 2 | 0 | 15 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 49 | 0 | 0 | 0 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| SRR6442698 | 0 | 9 | 537 | 2 | 0 | 0 | 4 | 0 | 8 | 4 | 0 | 0 | 2 | 1 | 2015 | 120 | 0 | 0 | 0 | 36 | 1 | 0 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 67 | 0 | 35 | 2 | 306 | 0 | 1 | 0 | 246 | 0 | 0 | 1 | 0 | 0 | 5 | 0 | 0 | 0 | 251 | 0 | 23 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 28 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 3 | 0 | 0 | 178 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 88 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 | 0 | 0 | 0 | 0 | 120 | 0 | 0 | 5 | 0 | 9 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 7 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| SRR6442699 | 0 | 581 | 0 | 0 | 1 | 0 | 2 | 3 | 6 | 0 | 8 | 0 | 1 | 0 | 2461 | 7 | 0 | 0 | 0 | 1400 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 236 | 0 | 0 | 0 | 575 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 168 | 0 | 1235 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1458 | 0 | 0 | 1208 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 92 | 0 | 0 | 21 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| SRR6442700 | 2 | 8300 | 3828 | 0 | 1 | 0 | 275 | 0 | 9 | 3557 | 8 | 0 | 3324 | 0 | 741 | 1045 | 413 | 6923 | 0 | 253 | 48 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 170 | 0 | 331 | 352 | 0 | 0 | 2913 | 0 | 0 | 2 | 967 | 164 | 0 | 0 | 146 | 361 | 0 | 0 | 0 | 0 | 0 | 2 | 28 | 0 | 17 | 0 | 0 | 0 | 1055 | 1 | 114 | 0 | 0 | 0 | 0 | 0 | 16 | 25 | 0 | 2 | 0 | 79 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 31 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 2 | 0 | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 | 1 | 1 | 4 | 197 | 0 | 0 | 39 | 160 | 0 | 0 | 0 | 0 | 28 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 36 | 0 | 0 | 0 |
| SRR6442701 | 0 | 80 | 526 | 2 | 1 | 88 | 0 | 25 | 249 | 2425 | 182 | 0 | 8 | 0 | 215 | 8 | 7742 | 2455 | 6 | 565 | 9 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 18 | 0 | 8 | 65 | 0 | 0 | 0 | 94 | 55 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 9 | 0 | 0 | 0 | 6 | 2550 | 0 | 690 | 1 | 3 | 0 | 0 | 77 | 0 | 85 | 40 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 107 | 2 | 0 | 0 | 0 | 0 | 0 | 72 | 0 | 0 | 0 | 0 | 40 | 101 | 3 | 0 | 108 | 0 | 0 | 0 | 0 | 0 | 241 | 0 | 0 | 0 | 0 | 0 | 0 | 12 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| SRR6442702 | 0 | 6 | 49 | 13341 | 3 | 0 | 3583 | 1 | 13608 | 5 | 0 | 0 | 15 | 460 | 3 | 272 | 19137 | 14544 | 0 | 18 | 2 | 0 | 1 | 1 | 3 | 90 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 15 | 0 | 1963 | 1 | 4 | 9 | 6 | 0 | 92 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 9 | 0 | 78 | 0 | 0 | 0 | 766 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 135 | 4 | 0 | 0 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 66 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 12 | 0 | 31 | 0 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
La tabla de cuentas relaciona el nombre de las taxa con las muestras y con el número de reads mapeadas en contra de ellas. Acá el número de reads es directamente proporcional al número de veces que se observa un taxon.
- El otro componente importante es la tabla de taxonomía.
# Esta es la tabla de taxonomía
head(tax_table(psd5)) %>%
kable(format = "html", col.names = colnames(tax_table(psd5))) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "320px")| Kingdom | Phylum | Class | Order | Family | Genus | |
|---|---|---|---|---|---|---|
| ASV1 | Bacteria | Proteobacteria | Gammaproteobacteria | Cardiobacteriales | Cardiobacteriaceae | NA |
| ASV2 | Bacteria | Bacteroidetes | Bacteroidia | Flavobacteriales | Flavobacteriaceae | NA |
| ASV3 | Bacteria | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | NA |
| ASV4 | Bacteria | Proteobacteria | Gammaproteobacteria | NA | NA | NA |
| ASV5 | Bacteria | Proteobacteria | Gammaproteobacteria | Xanthomonadales | Xanthomonadaceae | Stenotrophomonas |
| ASV6 | Bacteria | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | Moraxella |
La tabla de taxonomía relaciona el nombre de las taxa con el linaje taxonómico de éstas, i.e., vincula una variante de secuencia, o ASV, con los rangos taxonómicos desde Reino hasta Género o Especie dependiendo del nivel de resolución del análisis.
- Veamos ahora el otro componente esencial que es la metadata.
# Esta es la metadata
as(sample_data(psd5), "data.frame") -> metad
metad %>%
kable(format = "html", col.names = colnames(metad)) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "400px")| sample_ID | bioproject_accession | study | biosample_accession | experiment | run | SRA_Sample | geo_loc_name | collection_date | sample_type | species | common_name | AvgSpotLen | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| SRR6442697 | EMA4 | PRJNA428495 | SRP128093 | SAMN08292292 | SRX3533985 | SRR6442697 | SRS2809259 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442698 | EMA3 | PRJNA428495 | SRP128093 | SAMN08292291 | SRX3533984 | SRR6442698 | SRS2809258 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442699 | EMA2 | PRJNA428495 | SRP128093 | SAMN08292284 | SRX3533983 | SRR6442699 | SRS2809257 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442700 | EMA19 | PRJNA428495 | SRP128093 | SAMN08292283 | SRX3533982 | SRR6442700 | SRS2809256 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442701 | EMA21 | PRJNA428495 | SRP128093 | SAMN08292286 | SRX3533981 | SRR6442701 | SRS2809255 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442702 | EMA20 | PRJNA428495 | SRP128093 | SAMN08292285 | SRX3533980 | SRR6442702 | SRS2809254 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442703 | EMA23 | PRJNA428495 | SRP128093 | SAMN08292288 | SRX3533979 | SRR6442703 | SRS2809252 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442704 | EMA22 | PRJNA428495 | SRP128093 | SAMN08292287 | SRX3533978 | SRR6442704 | SRS2809253 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442705 | EMA25 | PRJNA428495 | SRP128093 | SAMN08292290 | SRX3533977 | SRR6442705 | SRS2809251 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442706 | EMA24 | PRJNA428495 | SRP128093 | SAMN08292289 | SRX3533976 | SRR6442706 | SRS2809250 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442708 | EMA18 | PRJNA428495 | SRP128093 | SAMN08292282 | SRX3533974 | SRR6442708 | SRS2809249 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442709 | EMA11 | PRJNA428495 | SRP128093 | SAMN08292275 | SRX3533973 | SRR6442709 | SRS2809248 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442710 | EMA12 | PRJNA428495 | SRP128093 | SAMN08292276 | SRX3533972 | SRR6442710 | SRS2809246 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442711 | EMA1 | PRJNA428495 | SRP128093 | SAMN08292273 | SRX3533971 | SRR6442711 | SRS2809245 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442712 | EMA10 | PRJNA428495 | SRP128093 | SAMN08292274 | SRX3533970 | SRR6442712 | SRS2809244 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442713 | EMA15 | PRJNA428495 | SRP128093 | SAMN08292279 | SRX3533969 | SRR6442713 | SRS2809243 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442714 | EMA16 | PRJNA428495 | SRP128093 | SAMN08292280 | SRX3533968 | SRR6442714 | SRS2809242 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442715 | EMA13 | PRJNA428495 | SRP128093 | SAMN08292277 | SRX3533967 | SRR6442715 | SRS2809241 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442716 | EMA14 | PRJNA428495 | SRP128093 | SAMN08292278 | SRX3533966 | SRR6442716 | SRS2809240 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442717 | F1 | PRJNA428495 | SRP128093 | SAMN08292272 | SRX3533965 | SRR6442717 | SRS2809239 | Chile: Chiloe | 2015 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442718 | CHIO5 | PRJNA428495 | SRP128093 | SAMN08292271 | SRX3533964 | SRR6442718 | SRS2809237 | Chile: Chiloe | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442719 | CHiO7 | PRJNA428495 | SRP128093 | SAMN08292270 | SRX3533963 | SRR6442719 | SRS2809238 | Chile: Chiloe | 2017 | skin | Eubalaena australis | southern right whale | 500 |
| SRR6442720 | CHiO6 | PRJNA428495 | SRP128093 | SAMN08292269 | SRX3533962 | SRR6442720 | SRS2809236 | Chile: Chiloe | 2017 | skin | Eubalaena australis | southern right whale | 501 |
| SRR6442721 | CHiO4 | PRJNA428495 | SRP128093 | SAMN08292268 | SRX3533961 | SRR6442721 | SRS2809235 | Chile: Chiloe | 2017 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442722 | CHiO3 | PRJNA428495 | SRP128093 | SAMN08292267 | SRX3533960 | SRR6442722 | SRS2809234 | Chile: Chiloe | 2017 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442723 | CHiO2 | PRJNA428495 | SRP128093 | SAMN08292266 | SRX3533959 | SRR6442723 | SRS2809232 | Chile: Chiloe | 2017 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442725 | F9 | PRJNA428495 | SRP128093 | SAMN08292264 | SRX3533957 | SRR6442725 | SRS2809231 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442726 | F8 | PRJNA428495 | SRP128093 | SAMN08292263 | SRX3533956 | SRR6442726 | SRS2809230 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442727 | F10 | PRJNA428495 | SRP128093 | SAMN08292242 | SRX3533955 | SRR6442727 | SRS2809229 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442731 | RNPH16 | PRJNA428495 | SRP128093 | SAMN08292238 | SRX3533951 | SRR6442731 | SRS2809225 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442732 | RNPH15 | PRJNA428495 | SRP128093 | SAMN08292237 | SRX3533950 | SRR6442732 | SRS2809224 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442733 | RNPH14 | PRJNA428495 | SRP128093 | SAMN08292236 | SRX3533949 | SRR6442733 | SRS2809223 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442734 | RNPH13 | PRJNA428495 | SRP128093 | SAMN08292235 | SRX3533948 | SRR6442734 | SRS2809222 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442735 | RNPH12 | PRJNA428495 | SRP128093 | SAMN08292234 | SRX3533947 | SRR6442735 | SRS2809221 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442736 | RNPH11 | PRJNA428495 | SRP128093 | SAMN08292233 | SRX3533946 | SRR6442736 | SRS2809220 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Balaenoptera physalus | fin whale | 501 |
| SRR6442737 | F6 | PRJNA428495 | SRP128093 | SAMN08292261 | SRX3533945 | SRR6442737 | SRS2809218 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442738 | F7 | PRJNA428495 | SRP128093 | SAMN08292262 | SRX3533944 | SRR6442738 | SRS2809219 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442739 | F39 | PRJNA428495 | SRP128093 | SAMN08292253 | SRX3533943 | SRR6442739 | SRS2809217 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442740 | F4 | PRJNA428495 | SRP128093 | SAMN08292254 | SRX3533942 | SRR6442740 | SRS2809216 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442741 | F40 | PRJNA428495 | SRP128093 | SAMN08292255 | SRX3533941 | SRR6442741 | SRS2809215 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442742 | F41 | PRJNA428495 | SRP128093 | SAMN08292256 | SRX3533940 | SRR6442742 | SRS2809214 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442743 | F42 | PRJNA428495 | SRP128093 | SAMN08292257 | SRX3533939 | SRR6442743 | SRS2809213 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442744 | F43 | PRJNA428495 | SRP128093 | SAMN08292258 | SRX3533938 | SRR6442744 | SRS2809212 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442745 | F44 | PRJNA428495 | SRP128093 | SAMN08292259 | SRX3533937 | SRR6442745 | SRS2809211 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442746 | F5 | PRJNA428495 | SRP128093 | SAMN08292260 | SRX3533936 | SRR6442746 | SRS2809210 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442747 | RNPH7 | PRJNA428495 | SRP128093 | SAMN08292328 | SRX3533935 | SRR6442747 | SRS2809209 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442748 | RNPH6 | PRJNA428495 | SRP128093 | SAMN08292327 | SRX3533934 | SRR6442748 | SRS2809208 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442749 | RNPH9 | PRJNA428495 | SRP128093 | SAMN08292330 | SRX3533933 | SRR6442749 | SRS2809207 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442750 | RNPH8 | PRJNA428495 | SRP128093 | SAMN08292329 | SRX3533932 | SRR6442750 | SRS2809206 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442751 | RNPH3 | PRJNA428495 | SRP128093 | SAMN08292324 | SRX3533931 | SRR6442751 | SRS2809205 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442752 | RNPH20 | PRJNA428495 | SRP128093 | SAMN08292323 | SRX3533930 | SRR6442752 | SRS2809204 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442753 | RNPH5 | PRJNA428495 | SRP128093 | SAMN08292326 | SRX3533929 | SRR6442753 | SRS2809203 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442754 | RNPH4 | PRJNA428495 | SRP128093 | SAMN08292325 | SRX3533928 | SRR6442754 | SRS2809202 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442755 | F16 | PRJNA428495 | SRP128093 | SAMN08292248 | SRX3533927 | SRR6442755 | SRS2809201 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442756 | F15 | PRJNA428495 | SRP128093 | SAMN08292247 | SRX3533926 | SRR6442756 | SRS2809199 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442757 | F18 | PRJNA428495 | SRP128093 | SAMN08292250 | SRX3533925 | SRR6442757 | SRS2809200 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442758 | F17 | PRJNA428495 | SRP128093 | SAMN08292249 | SRX3533924 | SRR6442758 | SRS2809198 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442759 | F12 | PRJNA428495 | SRP128093 | SAMN08292244 | SRX3533923 | SRR6442759 | SRS2809197 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442760 | F11 | PRJNA428495 | SRP128093 | SAMN08292243 | SRX3533922 | SRR6442760 | SRS2809196 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442761 | F14 | PRJNA428495 | SRP128093 | SAMN08292246 | SRX3533921 | SRR6442761 | SRS2809195 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442762 | F13 | PRJNA428495 | SRP128093 | SAMN08292245 | SRX3533920 | SRR6442762 | SRS2809193 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 |
| SRR6442763 | F3 | PRJNA428495 | SRP128093 | SAMN08292252 | SRX3533919 | SRR6442763 | SRS2809194 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442764 | F2 | PRJNA428495 | SRP128093 | SAMN08292251 | SRX3533918 | SRR6442764 | SRS2809192 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 |
| SRR6442765 | RNPH10 | PRJNA428495 | SRP128093 | SAMN08292321 | SRX3533917 | SRR6442765 | SRS2809191 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442766 | RNPH2 | PRJNA428495 | SRP128093 | SAMN08292322 | SRX3533916 | SRR6442766 | SRS2809190 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442767 | F50 | PRJNA428495 | SRP128093 | SAMN08292319 | SRX3533915 | SRR6442767 | SRS2809189 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442768 | RNPH1 | PRJNA428495 | SRP128093 | SAMN08292320 | SRX3533914 | SRR6442768 | SRS2809188 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442769 | F48 | PRJNA428495 | SRP128093 | SAMN08292317 | SRX3533913 | SRR6442769 | SRS2809187 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442770 | F49 | PRJNA428495 | SRP128093 | SAMN08292318 | SRX3533912 | SRR6442770 | SRS2809186 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442771 | F46 | PRJNA428495 | SRP128093 | SAMN08292315 | SRX3533911 | SRR6442771 | SRS2809185 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442774 | F45 | PRJNA428495 | SRP128093 | SAMN08292314 | SRX3533908 | SRR6442774 | SRS2809181 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442775 | F27 | PRJNA428495 | SRP128093 | SAMN08292306 | SRX3533907 | SRR6442775 | SRS2809180 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442776 | F26 | PRJNA428495 | SRP128093 | SAMN08292305 | SRX3533906 | SRR6442776 | SRS2809179 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442779 | F31 | PRJNA428495 | SRP128093 | SAMN08292310 | SRX3533903 | SRR6442779 | SRS2809176 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 499 |
| SRR6442780 | F30 | PRJNA428495 | SRP128093 | SAMN08292309 | SRX3533902 | SRR6442780 | SRS2809175 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442781 | F29 | PRJNA428495 | SRP128093 | SAMN08292308 | SRX3533901 | SRR6442781 | SRS2809174 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442782 | F28 | PRJNA428495 | SRP128093 | SAMN08292307 | SRX3533900 | SRR6442782 | SRS2809183 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442783 | F33 | PRJNA428495 | SRP128093 | SAMN08292312 | SRX3533899 | SRR6442783 | SRS2809173 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442784 | F32 | PRJNA428495 | SRP128093 | SAMN08292311 | SRX3533898 | SRR6442784 | SRS2809172 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442785 | F22 | PRJNA428495 | SRP128093 | SAMN08292301 | SRX3533897 | SRR6442785 | SRS2809171 | Chile: Estrecho_Magallanes | 2010 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442786 | F23 | PRJNA428495 | SRP128093 | SAMN08292302 | SRX3533896 | SRR6442786 | SRS2809170 | Chile: Estrecho_Magallanes | 2010 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442787 | EMA9 | PRJNA428495 | SRP128093 | SAMN08292297 | SRX3533895 | SRR6442787 | SRS2809169 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442788 | F19 | PRJNA428495 | SRP128093 | SAMN08292298 | SRX3533894 | SRR6442788 | SRS2809168 | Chile: Estrecho_Magallanes | 2010 | skin | Megaptera novaeangliae | humpback whale | 501 |
| SRR6442789 | F20 | PRJNA428495 | SRP128093 | SAMN08292299 | SRX3533893 | SRR6442789 | SRS2809167 | Chile: Estrecho_Magallanes | 2010 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442790 | F21 | PRJNA428495 | SRP128093 | SAMN08292300 | SRX3533892 | SRR6442790 | SRS2809166 | Chile: Estrecho_Magallanes | 2010 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442792 | EMA6 | PRJNA428495 | SRP128093 | SAMN08292294 | SRX3533890 | SRR6442792 | SRS2809164 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
| SRR6442794 | EMA8 | PRJNA428495 | SRP128093 | SAMN08292296 | SRX3533888 | SRR6442794 | SRS2809162 | Chile: Estrecho_Magallanes | 2017 | skin | Megaptera novaeangliae | humpback whale | 500 |
Finalmente, tenemos el árbol filogenético, que es opcional en phyloseq, que nos muestra las relaciones evolutivas entre las taxa de todas las muestras. Es opcional porque normalmente cuando hacemos shotgun metagenomics no contamos con un marcador universal y por lo tanto no hay filogenia.
- Podemos graficar simplemente la filogenia con la función
plot_tree.
# Esta es la filogenia asociada a las taxa en nuestro objeto phyloseq
plot_tree(psd5, method = "treeonly", ladderize = "left")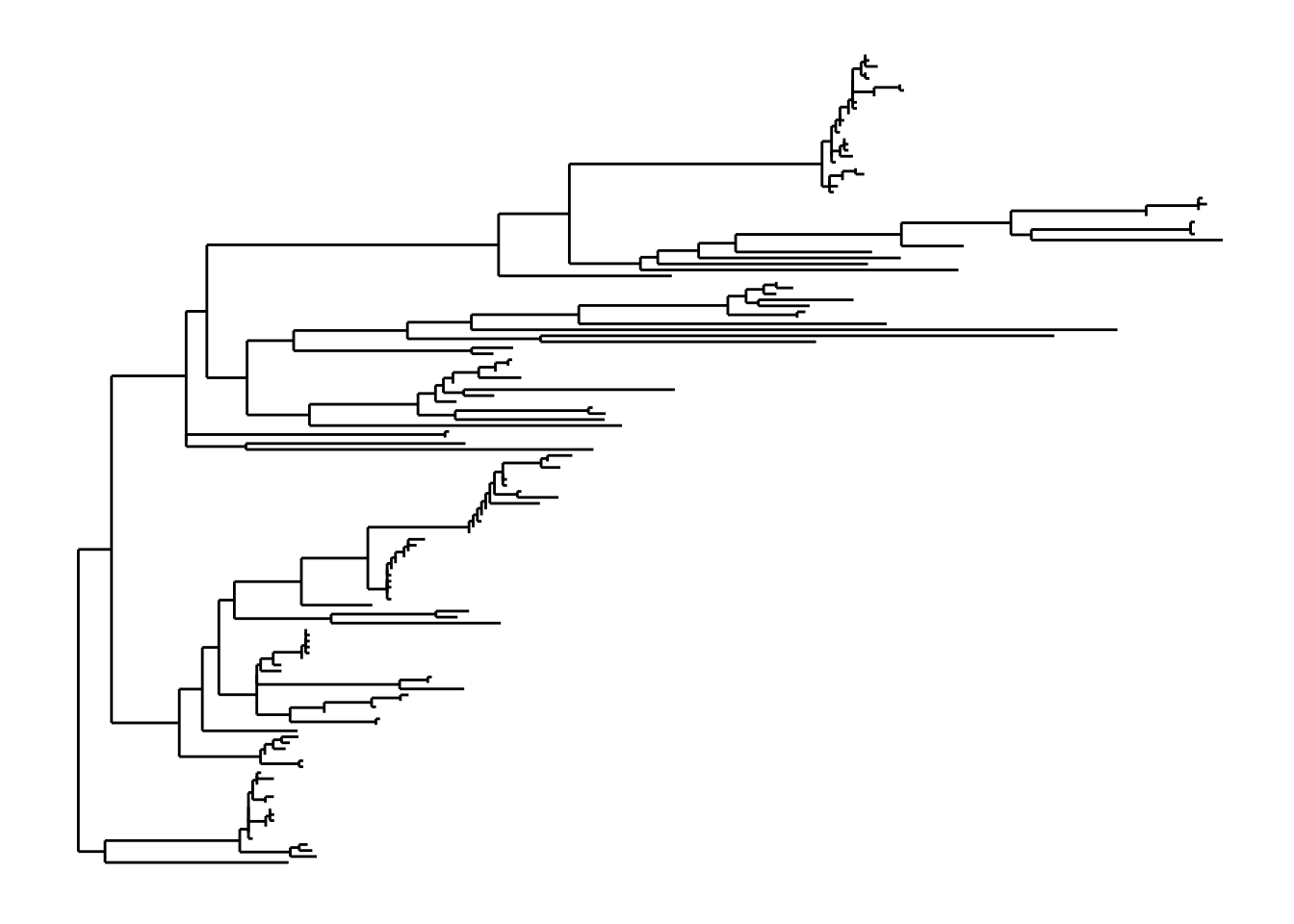
Ahora, el objeto phyloseq se ha vuelto una suerte de estándar en la industria ya que otros paquetes ahora usan esta estructura de datos para sus propias funciones. Uno de esos paquetes es microbiome y ampvis. Podemos fácilmente obtener un resumen global de nuestro objeto phyloseq usando la función summarize_phyloseq.
- Primero cargamos el paquete con
library(microbiome).
## [[1]]
## [1] "1] Min. number of reads = 1123"
##
## [[2]]
## [1] "2] Max. number of reads = 103541"
##
## [[3]]
## [1] "3] Total number of reads = 2606004"
##
## [[4]]
## [1] "4] Average number of reads = 29954.0689655172"
##
## [[5]]
## [1] "5] Median number of reads = 23576"
##
## [[6]]
## [1] "7] Sparsity = 0.679428668018932"
##
## [[7]]
## [1] "6] Any OTU sum to 1 or less? NO"
##
## [[8]]
## [1] "8] Number of singletons = 0"
##
## [[9]]
## [1] "9] Percent of OTUs that are singletons \n (i.e. exactly one read detected across all samples)0"
##
## [[10]]
## [1] "10] Number of sample variables are: 13"
##
## [[11]]
## [1] "sample_ID" "bioproject_accession" "study"
## [4] "biosample_accession" "experiment" "run"
## [7] "SRA_Sample" "geo_loc_name" "collection_date"
## [10] "sample_type" "species" "common_name"
## [13] "AvgSpotLen"Este comando nos muestra el mínimo y máximo de reads, número total y promedio de reads, etc. También muestra los encabezados de las columans en la tabla de metadata.
- Veamos ahora una tabla que mezcle metadata, taxonomíaa y abundancia del taxon más abundante de cada muestra.
df <- psmelt(psd5)
head(df) %>%
kable(format = "html", col.names = colnames(df)) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "400px")| OTU | Sample | Abundance | sample_ID | bioproject_accession | study | biosample_accession | experiment | run | SRA_Sample | geo_loc_name | collection_date | sample_type | species | common_name | AvgSpotLen | Kingdom | Phylum | Class | Order | Family | Genus | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 5 | ASV1 | SRR6442744 | 65451 | F43 | PRJNA428495 | SRP128093 | SAMN08292258 | SRX3533938 | SRR6442744 | SRS2809212 | Chile: Chiloe | 2016 | skin | Balaenoptera musculus | blue whale | 501 | Bacteria | Proteobacteria | Gammaproteobacteria | Cardiobacteriales | Cardiobacteriaceae | NA |
| 8009 | ASV6 | SRR6442747 | 57107 | RNPH7 | PRJNA428495 | SRP128093 | SAMN08292328 | SRX3533935 | SRR6442747 | SRS2809209 | Chile: Reserva_Nacional_Pinguino_de_Humboldt | 2017 | skin | Megaptera novaeangliae | humpback whale | 499 | Bacteria | Proteobacteria | Gammaproteobacteria | Pseudomonadales | Moraxellaceae | Moraxella |
| 39 | ASV1 | SRR6442738 | 46464 | F7 | PRJNA428495 | SRP128093 | SAMN08292262 | SRX3533944 | SRR6442738 | SRS2809219 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 501 | Bacteria | Proteobacteria | Gammaproteobacteria | Cardiobacteriales | Cardiobacteriaceae | NA |
| 3702 | ASV14 | SRR6442771 | 44378 | F46 | PRJNA428495 | SRP128093 | SAMN08292315 | SRX3533911 | SRR6442771 | SRS2809185 | Chile: Estrecho_Magallanes | 2016 | skin | Megaptera novaeangliae | humpback whale | 501 | Bacteria | Bacteroidetes | Bacteroidia | Flavobacteriales | Flavobacteriaceae | Tenacibaculum |
| 6139 | ASV4 | SRR6442763 | 42681 | F3 | PRJNA428495 | SRP128093 | SAMN08292252 | SRX3533919 | SRR6442763 | SRS2809194 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 | Bacteria | Proteobacteria | Gammaproteobacteria | NA | NA | NA |
| 4144 | ASV19 | SRR6442763 | 37149 | F3 | PRJNA428495 | SRP128093 | SAMN08292252 | SRX3533919 | SRR6442763 | SRS2809194 | Chile: Chiloe | 2015 | skin | Balaenoptera musculus | blue whale | 500 | Bacteria | Proteobacteria | Gammaproteobacteria | Cardiobacteriales | Cardiobacteriaceae | NA |
- También es importante tener una visión de cómo se distribuyen las muestras de acuerdo a la metadata. En este ejemplo, graficamos la frecuencia de muestras de acuerdo a la ubicación geográfica (geo_loc_name) y a la especie de ballena de donde la muestra fue obtenida (species).
## <environment: 0x7f8871c7e728># En formato tabla
res$data %>%
kable(format = "html", col.names = colnames(res$data), digits = 2) %>%
kable_styling() %>%
kableExtra::scroll_box(width = "100%", height = "300px")| Groups | Factor | n | pct | .group |
|---|---|---|---|---|
| Chile: Chiloe | Balaenoptera musculus | 26 | 86.67 | 1 |
| Chile: Chiloe | Eubalaena australis | 2 | 6.67 | 1 |
| Chile: Chiloe | Megaptera novaeangliae | 2 | 6.67 | 1 |
| Chile: Estrecho_Magallanes | Megaptera novaeangliae | 40 | 100.00 | 2 |
| Chile: Reserva_Nacional_Pinguino_de_Humboldt | Balaenoptera physalus | 6 | 35.29 | 3 |
| Chile: Reserva_Nacional_Pinguino_de_Humboldt | Megaptera novaeangliae | 11 | 64.71 | 3 |
Ahora veamos cómo podemos filtrar y hacer subsetting de un objeto phyloseq. Esto lo hacemos con tres grupos de funciones, i.e., filter, subset, y prune. Filtrar se refiere a filtrar según alguna regla lógica. Ya lo hicimos en la parte de control de calidad cuando llamamos la función filter_taxa(psd1, function(x) mean(x) > 1e-5, TRUE). Acá le pedíamos a la función filter_taxa que sobre el objeto psd5, calculara la media de read counts para cada taxa y si este resultado era menor que 1e-5, lo eliminara. Veamos un ejemplo diferente y filtremos según abundancia.
- Primero transformamos en abundancia relativa y luego filtramos.
# Transformamos las cuentas en porcentaje
psd5r = transform_sample_counts(psd5, function(x) x / sum(x) )
# Filtramos las taxa con una abundancia inferior al 1%
(psd5r.filtrado = filter_taxa(psd5r, function(x) sum(x) > 1, TRUE))## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 24 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 24 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 24 tips and 23 internal nodes ]¿Cuántas taxa permanecen en nuestro objeto phyloseq? Con una operación tan simple como la que acabamos de aplicar, nos damos cuenta que la mayoría de las taxa presentes en nuestras muestras están en muy baja abundancia.
- Ahora imaginemos la situación donde queremos filtrar nuestro objeto pero en función de un taxon en específico.
| Genus | |
|---|---|
| ASV1 | NA |
| ASV2 | NA |
| ASV3 | NA |
| ASV4 | NA |
| ASV5 | Stenotrophomonas |
| ASV6 | Moraxella |
| ASV7 | Tenacibaculum |
| ASV8 | Klebsiella |
| ASV9 | Tenacibaculum |
| ASV10 | NA |
| ASV11 | NA |
| ASV12 | Pseudomonas |
| ASV13 | NA |
| ASV15 | NA |
| ASV16 | Moraxella |
| ASV17 | Moraxella |
| ASV18 | NA |
| ASV19 | NA |
| ASV20 | NA |
| ASV22 | Pseudomonas |
| ASV23 | Tenacibaculum |
| ASV24 | Achromobacter |
| ASV25 | Catenococcus |
| ASV28 | Escherichia/Shigella |
# Ahora filtramos de acuerdo a _Moraxella_
(subset_taxa(psd5r.filtrado, Genus=="Moraxella") -> psd5r.filtrado.moraxella)## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 3 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 3 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 3 tips and 2 internal nodes ]# También podríamos todo lo que NO es _Moraxella_
(subset_taxa(psd5r.filtrado, Genus!="Moraxella") -> psd5r.filtrado.NoMoraxella)## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 10 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 10 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 10 tips and 9 internal nodes ]- Otra manera de filtrar un objeto
phyloseqes en base a algún atributo presente ensample_data. Por ejemplo, con estos datos uno podría querer estudiar el microbioma de las ballenas por separado. Para esto crearíamos tres objetosphyloseqa partir depsd5.
## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 26 samples ]
## sample_data() Sample Data: [ 26 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 6 samples ]
## sample_data() Sample Data: [ 6 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 53 samples ]
## sample_data() Sample Data: [ 53 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]- Alternativamente, podríamos decicir estudiar solo tres de las cuatro especies de ballenas que tenemos representadas en
psd5.
- El comando
prune_samples()también es muy usado ya que nos permite usar un vector con las muestras que queremos mantener (similar asubset_samples) o un vector lógico donde las muestras que queremos mantener son verdaderas.
# Primero seleccionamos solo el género _Moraxella_
subset_taxa(psd5, Genus=="Moraxella") -> psd5.moraxella
# Luego nos quedamos con las muestras que solo cumplen con la condición, i,e, que poseen una abundancia de _Moraxella_ de más de 5 reads
prune_samples(sample_sums(psd5.moraxella)>=5, psd5.moraxella) -> psd5.moraxella
# Y finalmente visualizamos los resultados mapeados en el árbol filogenético
plot_tree(psd5.moraxella, color="species", shape="Family", label.tips="Genus", size="abundance")
Inmediatamente podemos apreciar que la distribución de Moraxella es mayor en ballena jorobada que en las otras dos especies, azul y fin.
- Otra situación muy común ocurre cuando queremos remover contaminantes u otras taxa no deseadas. Esto se puede hacer fácilmente con el comando
prune_taxa.
# Primero definimos las taxa que no queremos
badTaxa = c("ASV134", "ASV104", "ASV68")
# Creamos una lista con todos los nombres de los taxa presentes en el objeto `psd5`
allTaxa = taxa_names(psd5)
# Nos quedamos con la diferencia entre badTaxa y allTaxa
keepTaxa <- allTaxa[!(allTaxa %in% badTaxa)]
#Ejecutamos `prune_taxa` sobre psd5
(psd5.prune = prune_taxa(keepTaxa, psd5))## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 133 taxa and 85 samples ]
## sample_data() Sample Data: [ 85 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 133 taxa by 6 taxonomic ranks ]
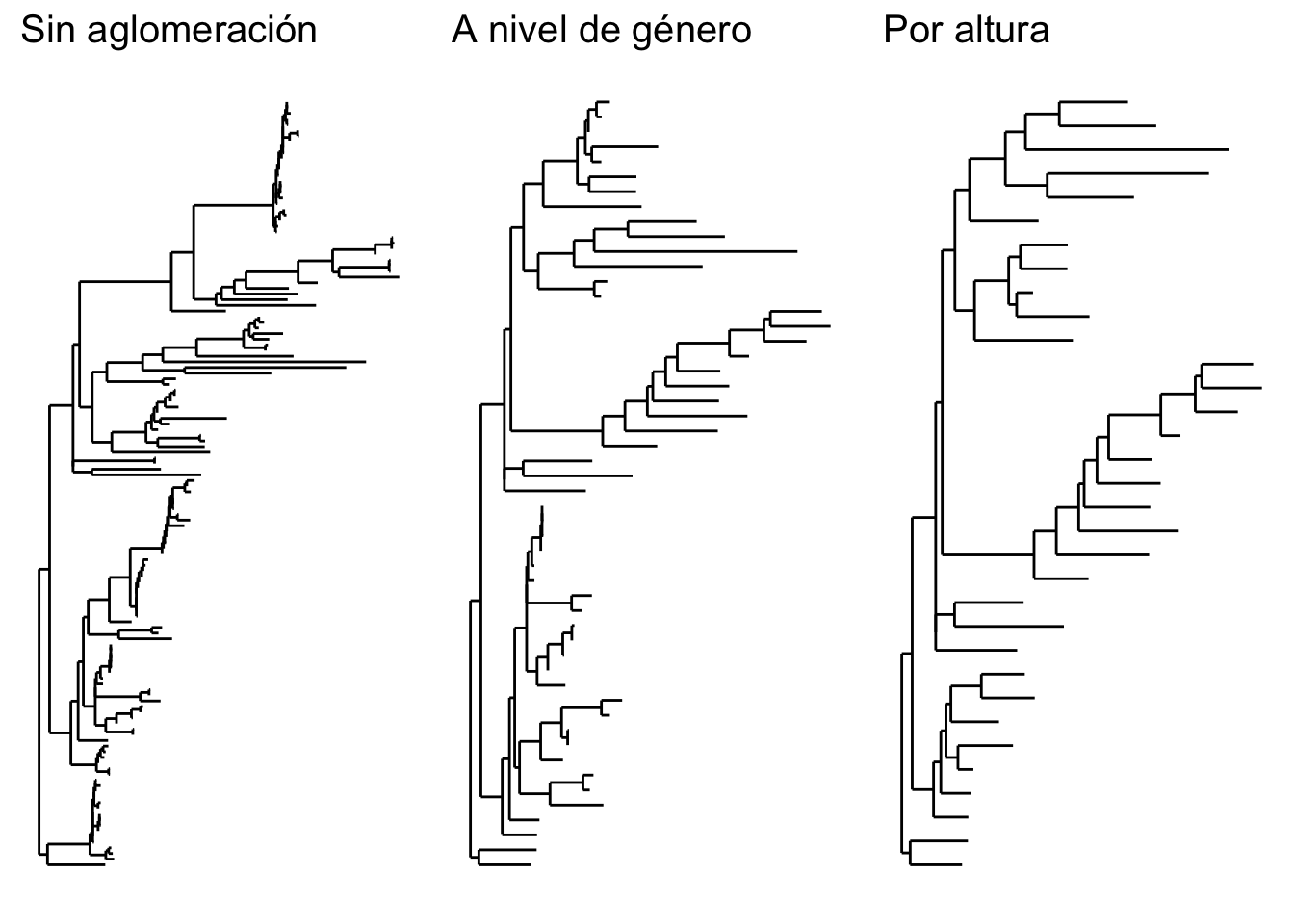
## phy_tree() Phylogenetic Tree: [ 133 tips and 132 internal nodes ]Para finalizar esta sección, un par de funciones muy útiles en phyloseq son tax_glom() y tip_glom(). Ambas funciones tratan de agrupar o aglomerar un objeto de acuerdo a alguna propiedad, de esta manera simplificándolo. Por ejemplo, es muy probable que uno tenga varias ASVs del mismo género ya que si bien a nivel de secuencia son diferentes, estas corresponden al mismo género. En cierta forma ya lo vimos cuando seleccionamos el género Moraxella. El objeto resultante tenía ocho taxa, todas ellas Moraxella.
- Para hacer visualizaciones y otros análisis puede ser conveniente colapsar o aglomerar estas secuencias del mismo género u otro rango taxonómico. Al mismo tiempo,
tip_glomrealiza una función similar pero basándose en una “altura” arbitraria en el árbol filogenético.
# Primero aglomeramos por género
psd5.genus = tax_glom(psd5, "Genus", NArm = FALSE)
# Luego por altura en el árbol filogenético
h1 = 0.4
psd5.tip = tip_glom(psd5, h = h1)
# Grafiquemos una comparación para visualizar las diferencias
multiPlotTitleTextSize = 15
p2tree = plot_tree(psd5, method = "treeonly",
ladderize = "left",
title = "Sin aglomeración") +
theme(plot.title = element_text(size = multiPlotTitleTextSize))
p3tree = plot_tree(psd5.genus, method = "treeonly",
ladderize = "left", title = "A nivel de género") +
theme(plot.title = element_text(size = multiPlotTitleTextSize))
p4tree = plot_tree(psd5.tip, method = "treeonly",
ladderize = "left", title = "Por altura") +
theme(plot.title = element_text(size = multiPlotTitleTextSize))
# Graficamos los árboles juntos
grid.arrange(nrow = 1, p2tree, p3tree, p4tree)